Kaggle Wyzwanie - Titanic
Wstęp
Witam wszystkich chętnych którzy chcieli by spróbować wyzwania Kaggle - Titanic. Jest to pierwsze zadanie z którym stykają się wszyscy na Kaggle zaraz po rejestracji.
Wyzwanie jest zaplanowane na 2 tygodnie od poniedziałku (09-07-2018) do następnego poniedziałku (23-07-2018). Jeśli termin już minął ta stroną będzie cały czas na Github tak żeby mieć możliwość przystąpić później.
Będzie on zarówno w R jak i Python tak żeby każdy nie był pokrzywdzony. Jest on podstawowy, dlatego można pytać o wszystko (od instalacji pythona, co to jest pip, po statystykę, itp..).
Planuje żeby w ciągu tygodnia robić 3 omówienia (w poniedziałek, środę i piątek) rozłożone na dwa tygodnie tak żeby każdy mógł mieć czas na przemyślenia, napisanie kodu i jeszcze pytania.
- (poniedziałek) Opis problemu, Importowanie danych i podstawowe informacje
- (środa) Wykresy i wyszukiwanie danych
- (piątek) Wypełnianie pustych danych i normalizacja Danych
- (poniedziałek) Klasyfikacja - algorytm
- (środa) Poprawianie Modelu
- (piątek) Podsumowanie
Podczas każdego omówienia postaram się opisać (github) co się dzieje i podać rozwiązanie dla każdego języka. W razie jakilkowiek pytań będzie można zadawać na slacku. Prosze mieć na uwadzę że też się uczę części rzeczy (zwłaszcza R), tak więc jak będzie jakiś błąd, niezrozumienie z mojej strony albo uwagi, proszę mnie o tym poinformować a ja to postaram się poprawić 😃
Wyzwanie można śledzić na grupie: https://www.facebook.com/groups/1733307126704677/
Osoby chętne, proszę dołączyć do slacka: https://kagglepolska.slack.com (link na grupie) A pytania można zadawać na grupie pod #titanic
Przed warto sobie zainstalować środowiska. Do pythona polecam Anaconda które zawiera Pythona oraz edytor Spyder:
https://www.anaconda.com/download/
Do R Edytor: Rstudio https://www.rstudio.com/products/rstudio/download/ oraz interpretator: Microsoft R Open https://mran.microsoft.com/open
Link do Titanic na kaggle: https://www.kaggle.com/c/titanic
Zaczynamy od poniedziałku, i życzę powodzenia 😃
Titanic, Wstęp
Jak można wyczytać, głównym zamiarem jest sklasyfikowanie pasażerów pod względem przeżycia podczas zatonięcia Tytanica które to miało miejsce w 15 kwietnia 1912. Jest to przykład binarnej klasyfikacji w której na podstawie danych wejściowych (takich jak klasa, numer biletu, płeć itp..) mamy wywnioskować czy osoba ta przeżyła wypadek.
Nie licząc żę był to zwykły łut szczescia można wyobrażić sobie kilka czynników które rzeczywiście miały na to wpływ. Do nich można zaliczyć:
- Jak blisko ich kajuty były łodzi ratunkowych
- W jakim wieku byli, np: młodsze osoby albo kobiety z dziećmi miały większe prawdopobieństwo dostania się do kajuty.
- Osoby mające droższe bilety a więc wyżej sytuowane mogli mieć jakieś przywileje
I tutaj to badanie ma za zadanie wyliczyć to powiązanie. Na początku warto zobaczyć sobie dane wejściowe. Na wstępie mamy takie oto kolumny:
| Variable | |
|---|---|
| Zmienna (Variable) | Opis |
| Survived | Czy dana osoba przeżyła. 0 - Nie, 1 - Tak. Ta wartość pojawia się tylko w test |
| PClass | Klasa socio-economic (SEC), 1- Upper, 2 - Middle, 3rd - Lower |
| Name | Imi i nazwisko oddzielone przecinkiem |
| Sex | Płeć, ‘male’ - męzczyzna , ‘female’- kobieta |
| Age | wiek |
| SibSp | (siblink) liczba rodzeństwa (brat i siostra) / (spouses) małżonków na pokładzie |
| Parch | iczba rodziców / dziecie na pokładzie |
| Fare | opłata za bilet |
| cabin | numer kabiny |
| embarked | Port źródłowy /załadunkowy (C - Cherbourk, Q - Quenstown, S - Southampton) |
Pełny wygląd klas można zobaczyć na stronie: https://www.encyclopedia-titanica.org/class-gender-titanic-disaster-1912~chapter-2.html
Przeglądając pobieżnie można zobaczyć że nie wszystkie dane są wypełnione, więc trzeba je w pewnym momencie wypełnić.
Python/ Wstęp
Zaczynając prace z Kaggle mamy tak naprawdę 3 możliwości:
- Możemy ściągnąć dane train i test i działać na nim lokalnie
- Możemy utworzyć Kernel i wysłać skrypt który rozwiąże zadanie
- Możemy utworzyć Kernel i za pomocą edytora “Jupyter Notebooks”nie tylko wykonać wszystkie operacje ale także dodać w wygodnym edytorze MarkDown własne przypisy czy wykresy.
Ja wybrałem Markdown bo można na bieżąco kontrolować co się dzieje. Jeśli chcesz utworzyć nowy kernel na stronie https://www.kaggle.com/c/titanic wybieramy Kernels -> New Kernel -> Notebook u góry po prawej możemy zmienić język z Python (który jest domyślny) na R.
Python/ Podstawowe Operacje
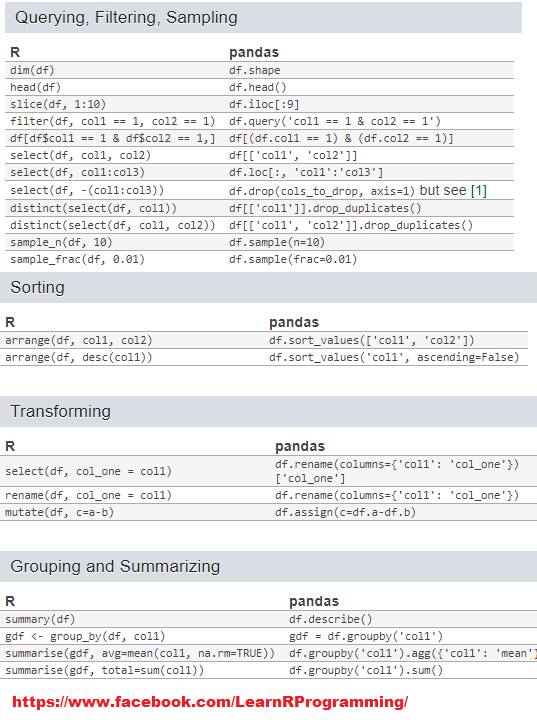
Do podstawowych operacji pandas dobrze popatrzeć na cheatsheet:
http://pandas.pydata.org/Pandas_Cheat_Sheet.pdf
Krótkie porównanie znalezione na facebook-u.
Biblioteki
Pierwsze co pozostaje to zaimportowanie najważniejszych bibliotek które przydadzą się do operacji na danych.
import numpy as np # linear algebra
import pandas as pd # data processing, CSV file I/O (e.g. pd.read_csv)
from subprocess import check_output
print(check_output(["ls", "../input"]).decode("utf8"))
- numpy - podstawowy biblioteka do algebry liniowej
- pandas - biblioteka dzięki której można używać powszechne struktury data.frame bardzo pomocne przy obliczeniach
pd.read_csv(name,header, index_col)
Do importu danych służy polecenia pd.read_csv() z biblioteki pandas. Bibliotek ta zwraca nam automatycznie obiekt DataFrame. Ten obiekt po pierwsze jest wygodniejszy do obliczeń typowych danych które są w formie dwuwymiarowej (wiersze i kolumny) a po drugie szybszy.
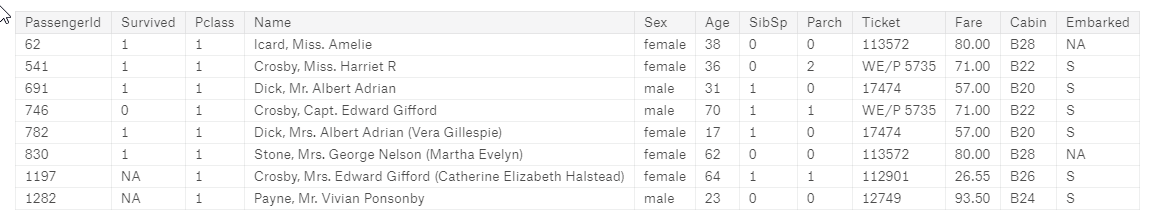
- header - 0 wskazuje że zerowy wiersze będzie nam wskazywał nazwy kolumn
- index_col - Wskazuje gdzie się znajduje kolumna z id (tutaj PassengerID)
# Load the data, we set that index_col is the first column, therefore there will be standard index start from 0 for each data.
train_df = pd.read_csv('../input/train.csv', header=0,index_col=0)
test_df = pd.read_csv('../input/test.csv', header=0,index_col=0)
pd.concat(a,b)
To polecenie na początku łączy nam oba obiekty po wierszach. Analizę danych powinniśmy robić na całym obszarze, bo może się zdarzyć że np: w train kolumna ma wszystkie pola wypełnione ale w tests już nie.
full = pd.concat([train_df , test_df]) # concatenate two dataframes
df.info()
Pokazuje podstawowe informacje obiektu, pozwala się rozejrzeć co za dane są w posczególnych kolumnach.
full.info() # info about dataframe
<class 'pandas.core.frame.DataFrame'>
Int64Index: 1309 entries, 1 to 1309
Data columns (total 11 columns):
Age 1046 non-null float64
Cabin 295 non-null object
Embarked 1307 non-null object
Fare 1308 non-null float64
Name 1309 non-null object
Parch 1309 non-null int64
Pclass 1309 non-null int64
Sex 1309 non-null object
SibSp 1309 non-null int64
Survived 891 non-null float64
Ticket 1309 non-null object
dtypes: float64(3), int64(3), object(5)
memory usage: 122.7+ KB
- Ponieważ 1309 to jest łączna liczba wierszy, to tam gdzie znajduje się ich mniej (poza Survived które nie występuje w test_df) znajdują się wartości które nie są wypełniona.
df.head()
Pokazuje pierwsze wiersze danych i można się spostrzec że Cabin, Embarked, Name, Sex oraz Ticket które .info() są jako object tak naprawdę to tekst.
full.head()
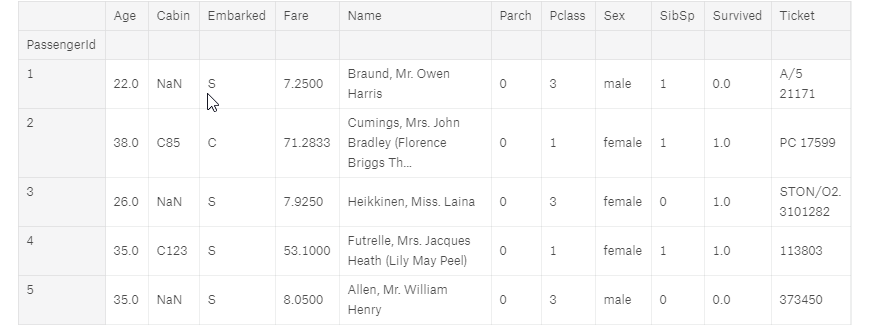
Wybieranie kolumn
# import matplotlib.pyplot as plt
full[["Age","Pclass"]][5:30]
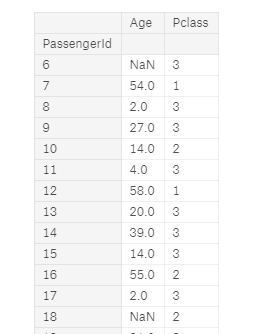
Filtrowanie
Przykłady wybierania wierszy:
- Jak w pythonie do każdej listy można wybierać przez indeks
full[:10] # Pierwszych 10 elementów
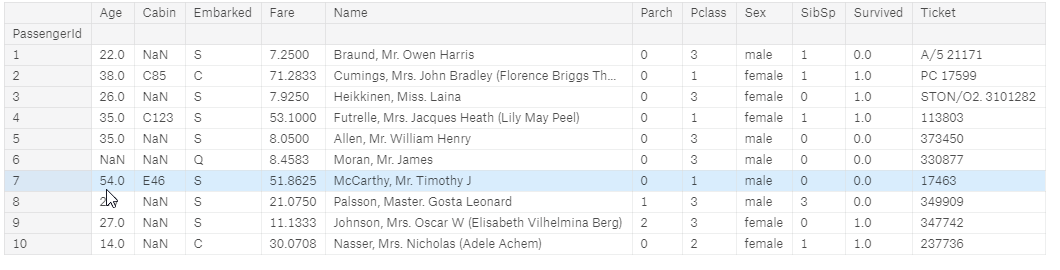
- 5 ostatnich elementów
full[-5:]
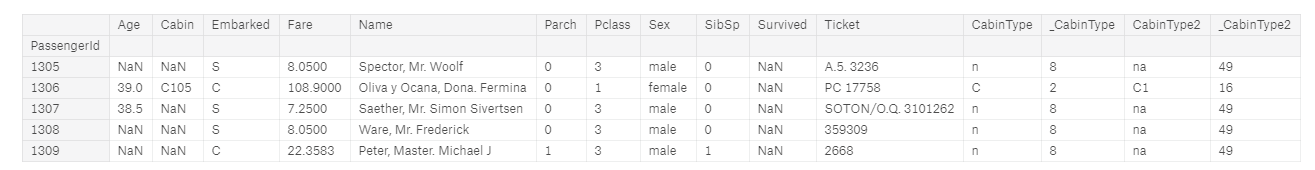
- Konkretnie od konkretnego wiersza idąc co 2, 891 - to pierwszy wiersz z bazy test_df
SURV = 891
full[SURV:SURV+10:2] # Like in regular Python you can get to the Item by Index
- filtrowanie po kolumnie
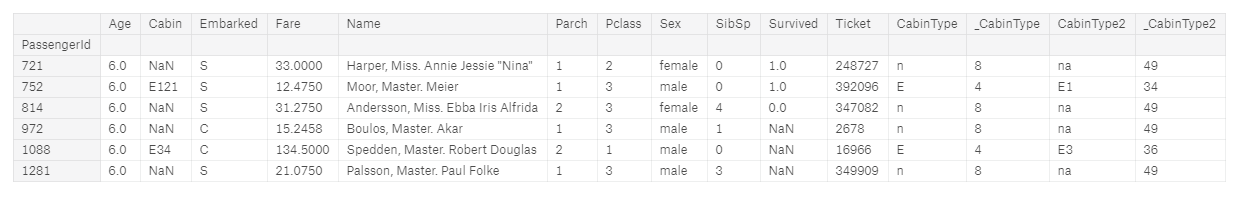
full[(full['Age'] > 5.0) & (full['Age'] < 7.0 ) ] #filter data by columns
- Filtrowanie po tekście
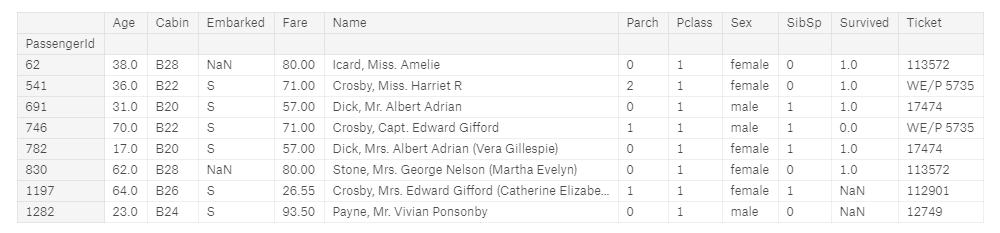
full[(full['Cabin'].str.contains('B2',na=False)) ] #filter data by columns
- filtrowanie po pustych wartościach
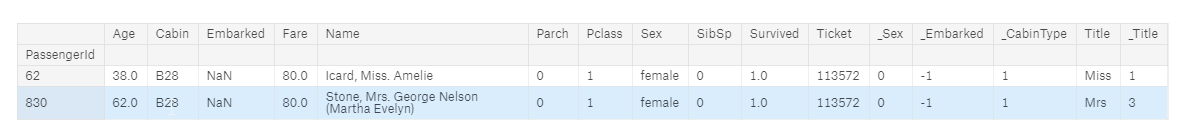
full[full['Embarked'].isnull()]
df.isnull()
Najważniejsze jest znalezielenie pustych wierszy. .isnull() zwraca nam cały zbiór z wartościami False,True czy jest pusta, .sum grupuje je dla wszystkich kolumn.
full.isnull().sum() # Check with alues are empty
Age 263
Cabin 1014
Embarked 2
Fare 1
Name 0
Parch 0
Pclass 0
Sex 0
SibSp 0
Survived 418
Ticket 0
CabinType 0
_CabinType 0
CabinType2 0
_CabinType2 0
dtype: int64
- Wartości Age,Cabin, Embarked, Fare, trzeba będzie uzupełnić danymi przed dalszym przetwarzaniem
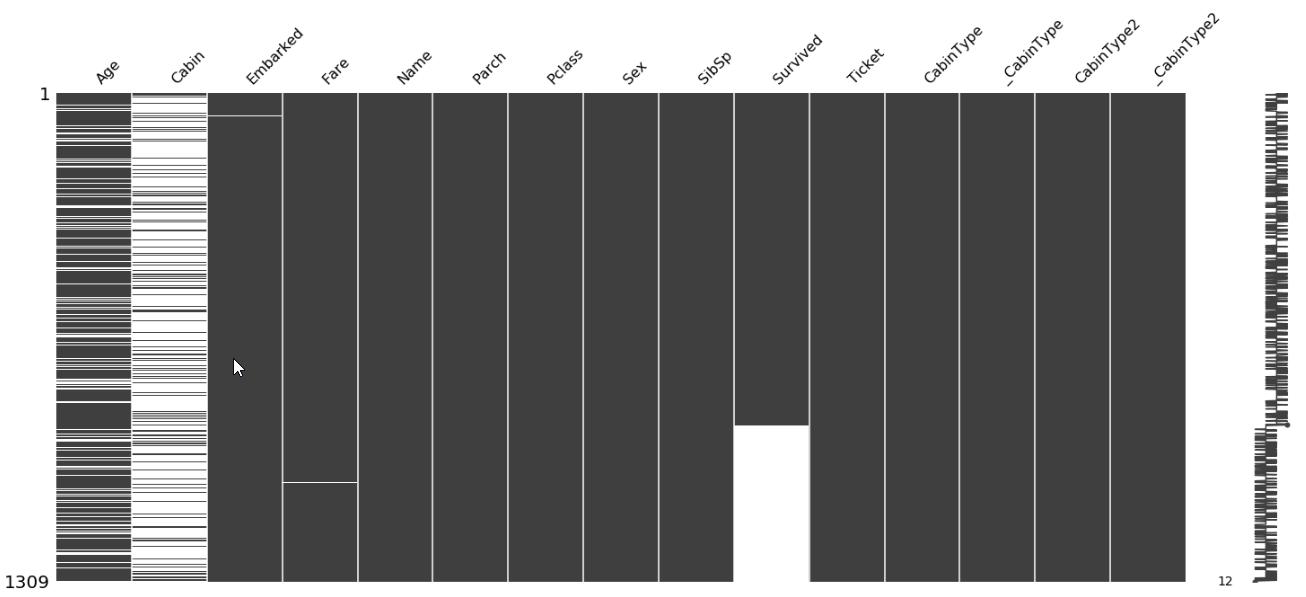
- Istnieje także wykres który pokazuje puste wartości
#Missing values in the plot
import missingno as msno
msno.matrix(full)
groupby()
Dane też można grupować by zobaczyć jak wyglądają zmienne na poszczególne grupy (np: sumujemy sobie ilość osób które przeżyły w podziale na klasy i płeć)
train_df.groupby(['Pclass','Sex'])['Survived'].sum() # grouping data
Pclass Sex
1 female 91
male 45
2 female 70
male 17
3 female 72
male 47
Name: Survived, dtype: int64
Python/ Wykresy i Dane
Tak naprawdę pod tym pojęciem chciałem napisać w jaki sposób można dodać kolumny zawierające dodatkowe informacje które kryją się w surowych danych ponieważ może się zdarzyć że:
- Jakieś informacje znajdują się w tekście a ponieważ znajdują się tam także inne informacje to algorytm nie wykryje że dana zmienna ma istotny wpływ
- Niektóre dane są zbyt rozdrobnione (np: wiek) przez co zaburzają obraz całego modelu. Przecięz podczas próby uratowania się nikt nie pytał dokładnie o wiek, więc można podzielić je na mniejsze grupy typu małe dzieci, dorośli itp..
- Część danych jest w postaci tekstowej, i trzeba je skategoryzować.
Python/ Dane
Kategoryzacja
- Pierwszym problemem w danych są dane tekstowe którze trzeba skategoryzować. W tym wypadku można użyć pandas.Categorical()
full['_Sex'] = pd.Categorical(full.Sex).codes
full['_Embarked'] = pd.Categorical(full.Embarked).codes
W nowych kolumnach pojawią wartości 0 1
Kategoryzacja kolumny Cabin
- Także tą kolumnę można skategoryzować. Można zauważyć że pierwsza litera może mieć jakieś znaczenie. Nie wszystkie dane są wypełnione,
full['_CabinType'] = pd.Categorical(full['Cabin'].astype(str).str[0]).codes
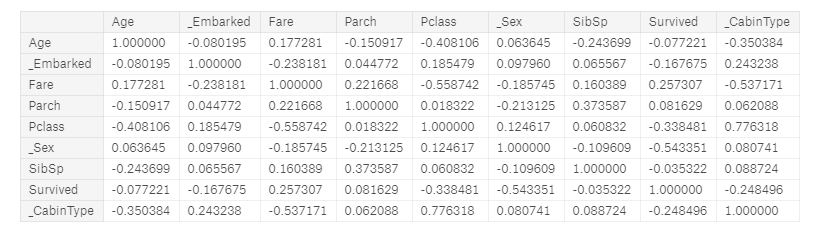
Korelacja
- Korelacja przydaje się do sprawdzenia zależności w kolumnach
- Korelacja wynosząca 1.0 oznacza pełną dodanie powiązanie (Jest na przekątnych),
- Korelacja wynosząca -1.0 oznacza odwrotne powiązanie
- Im bliższe 0 tym korelacja jest mniejsza
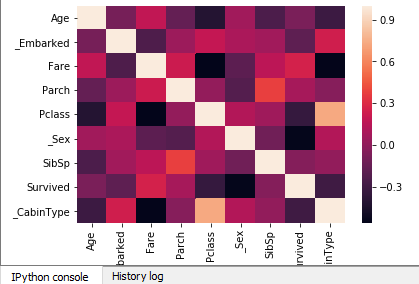
Z poniższego można wywnioskować że największy wpływ na przeżycie może mieć Pclass, _Sex, Fare, oraz _ CabinType. Musieliśmy wcześniej skategoryzować kolumnu ponieważ korelacji nie można sprawdzić na tekście.
cols = ['Age','_Embarked','Fare','Parch','Pclass','_Sex','SibSp','Survived','_CabinType']
full[cols].corr()
Tytuł - Pattern
- Warto sobie wyciągnąć tytuł z imienia i nazwiska czyli kolumny Name. Do tego przydaje się regular expression
pat = r",\s([^ .]+)\.?\s+"
full['Title'] = full['Name'].str.extract(pat,expand=True)[0]
full.groupby('Title')['Title'].count()
Title
Capt 1
Col 4
Don 1
Dona 1
Dr 8
Jonkheer 1
Lady 1
Major 2
Master 61
Miss 260
Mlle 2
Mme 1
Mr 757
Mrs 197
Ms 2
Rev 8
Sir 1
the 1
Name: Title, dtype: int64
Najwięcej jest Miss, Master, Mr, oraz Mrs. Więc dla lepszego obrazu można zgrupować część danych.
full.loc[full['Title'].isin(['Mlle','Ms','Lady']),'Title'] = 'Miss'
full.loc[full['Title'].isin(['Mme']),'Title'] = 'Mrs'
full.loc[full['Title'].isin(['Sir']),'Title'] = 'Mr'
full.loc[~full['Title'].isin(['Miss','Master','Mr','Mrs']),'Title'] = 'Other' # NOT IN
full['_Title'] = pd.Categorical(full.Title).codes
full.groupby('Title')['Title'].count()
Title
Master 61
Miss 263
Mr 757
Mrs 199
Other 29
Name: Title, dtype: int64
- Od razu zmniejszyła się ilość kategorii
TicketCounts
full['TicketCounts'] = full.groupby(['Ticket'])['Ticket'].transform('count')
- Wyciągnałem tak też nową kolumnę TicketCounts które oznacza ile osób ma ten sam numer biletu.
Python/ Wykresy
W pythonie do podstawowych wykresów głównie się używa matplotlib, seaborn oraz ggplot (bazujące na ggplot2 z R-a).
Histogram
- Podstawowym wykresem jest histogram czyli rozłożenie wartości według częstości występowania.
full['Age'].hist();
Od razu widać żę najwięcej jest osób w przedziale od 20 do 30 roku życia.
- Ale możemy chcieć też większy podział albo przefiltrowane na tylko te które przeżyły
full[full['Survived']==1]['Age'].hist(bins=30)
- Można też nałożyć na siebie histogramy (za pomocą plt z matplotlib). Tworzymy dwa histogramy (plt_all i plt_survived), dodajemy legendę i wyświetlamy.
import matplotlib
import matplotlib.pyplot as plt
plt_all = plt.hist(full['Age'],bins = 30, range = [0,100],label='all')
plt_survived =plt.hist(full[full['Survived']==1]['Age'], bins = 30, range = [0,100],label='Survived')
plt.legend()
plt.show()
Zauważyć można żę młodsi mają większą szansę przeżycia (pomarańczowa pokrywa połowę wartości albo większość dla od 0 do 10)
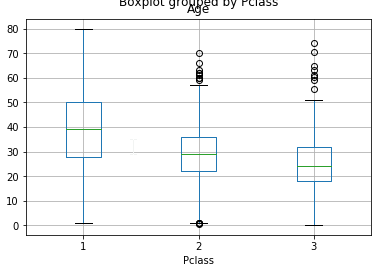
Boxplot
Zawiera on dużo informacji więc lepiej doczytać co oznaczają poszczególne wartości
https://www.wellbeingatschool.org.nz/information-sheet/understanding-and-interpreting-box-plots
full.boxplot(column='Age')
full.boxplot(column='Age',by='Pclass')
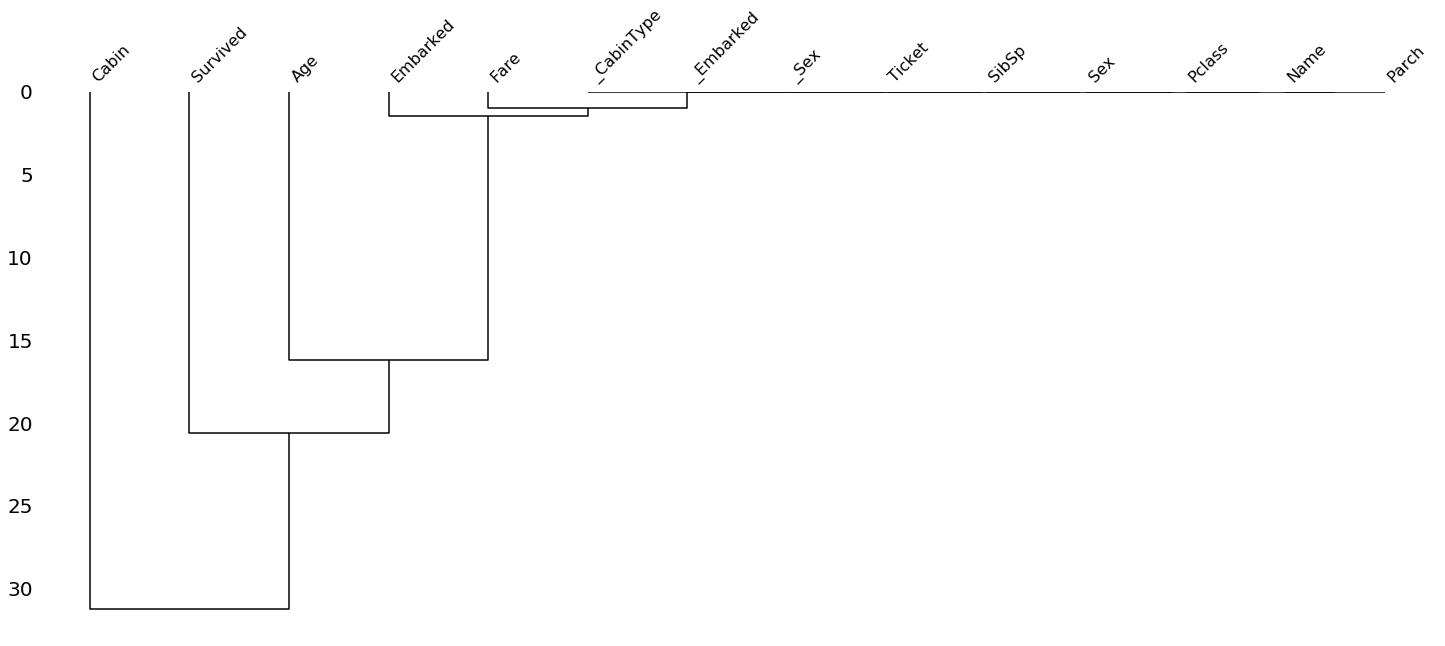
Dendrogram
- Jednym z ciekawszych wykresów jest dendogram, który pokazuje zależności danych w postaci drzewa
Ten wykres można potraktować jako ciekawostkę, jedyne co można odczytać to to że Cabin i Age są najbardziej wpływowymi kolumnami.
import missingno as msno
msno.dendrogram(full)
Heatmap / Correlation Map
- Heatmap pozwala zobaczyć zależności z korelacji. Ujemne korelacje są na ciemniejsz, a dodanie na coraz jaśniejsze.
cols = ['Age','_Embarked','Fare','Parch','Pclass','_Sex','SibSp','Survived','_CabinType']
corr = full[cols].corr()
import seaborn as sns
sns.heatmap(corr)
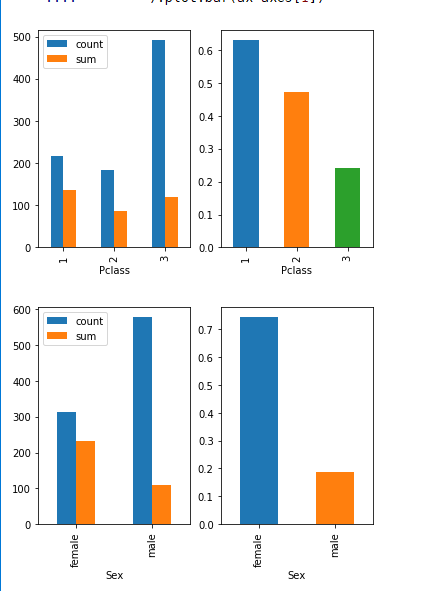
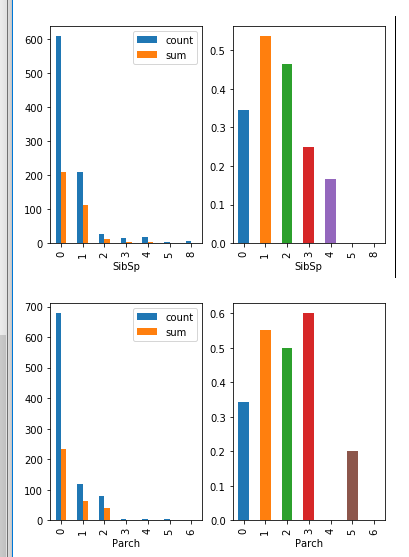
Wykresy zbiorowe
- Wykresy można grupować przez sublots() - podajemy ilość kolumn i wierszy. Np, ja mogę łatwo zobaczyć stosunek ilości przeżytych do całości.
for column in ['Pclass','Sex','SibSp','Parch','Embarked']:
fig, axes = plt.subplots(nrows=1, ncols=2)
(train_df
.groupby(column)['Survived']
.agg(['count','sum'])
).plot.bar(ax=axes[0])
(train_df
.groupby(column)['Survived']
.mean()
).plot.bar(ax=axes[1])
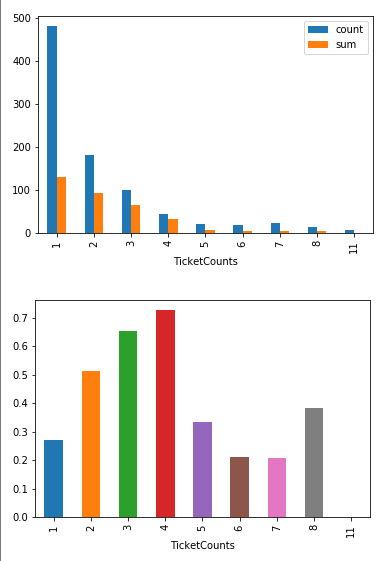
- Dodatkowo możemy obliczyć jakie jest prawdopodobieństwo przeżycia w zależności od osób które mają ten sam numer biletu.
(full
.groupby('TicketCounts')['Survived']
.agg(['count','sum'])
).plot.bar()
plt.show()
(full
.groupby('TicketCounts')['Survived']
.mean()
).plot.bar()
plt.show()
Python/ Uzupełnianie pustych wartości
Jak zauważyliśmy wiele wartości jest pustych i dochodzimy do ważnego momentu kiedy te wartości trzeba uzupełnić pewnymi danymi. Można to zrobić na kilka sposobów:
- Usunąć wiersze które posiadają puste wartości, ale w tym wypadku w modelu pozostałoby zbyt mało danych do analiz
- Usunąć kolumny które posiadają puste wartości, ale część kolumn ma istotny wpływ na wynik (np: Age)
- Wstawić wartość zerową - np: 0, ale może to zaburzyć bardzo póżniejszy model.
- Interpolate Value - Wstawiś średnią, medianę ze zbioru. Wtedy nie zaburza aż tak bardzo wyników ale nie jest tak dokładna
- Forward Fill/Backward Fill - Wtedy wypełniane są wartości na podstawie tego co było we wcześniejszym wierszu idąc od początku (Forward) lub idąc od końca co było w następnym wierszu (Backward).
Przykład R:
data <- data.frame(country=c("AUT", "AUT", "AUT", "AUT", "GER", "GER", "GER", "GER", "GER"), value=c(NA, 5, NA, NA, NA, NA, 7, NA, NA))
require(zoo)
na.locf(data, na.rm=FALSE) # Forward
na.locf(data, fromLast = TRUE, na.rm=FALSE) #Backward
Przykład Python:
data = pd.DataFrame({
'country': ["AUT", "AUT", "AUT", "AUT", "GER", "GER", "GER", "GER", "GER"],
'value:': [np.nan, 5, np.nan, np.nan, np.nan, np.nan, 7, np.nan, np.nan]
})
data.fillna(method='ffill') #forward
data.fillna(method='bfill') #backward
| Przed | Forward | Backward |
|---|---|---|
- Impute value - Możemy wyliczyć metodami statystycznymi albo innymi jak dane mają zostać uzupełnione
Embarked
- Embarked jest tylko dwóch i pochodzą z tej samej kabiny, w dodatku to dwie osoby które zapłaciły tyle samo za bilet
full[full['Embarked'].isnull()]
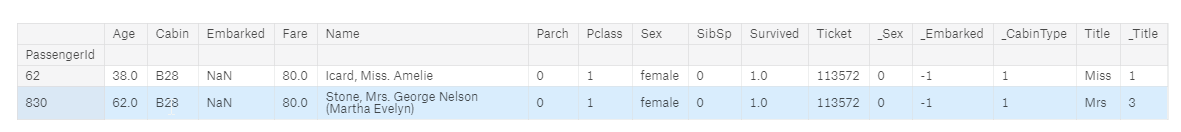
Z tego można wysnuć wniosek że można Embarked wypełnić wartościami z wierwszy o zbliżonych wartościach. Najpierw sprawdzimy korelacji wartości _Embarked od czego ona najbardziej zależy. Według danych zależy od _CabinType i Pclass
abs(full[cols].corr()['_Embarked']).sort_values(ascending=False)
_Embarked 1.000000
_CabinType 0.242810
Fare 0.241442
Pclass 0.192867
Survived 0.176509
_Sex 0.104818
Age 0.089292
SibSp 0.067802
Parch 0.046957
Name: _Embarked, dtype: float64
Wyświetliłem sobie iloe kosztował bilet 1 klasy dla kabin ‘B’ _CabinType = 1
val = full[ (full['Pclass'] == 1)
& (full['_CabinType'] == 1)
][['Fare','Embarked']];
val.groupby(['Embarked' ])['Fare'].agg(['count','min','max','mean','median'])
Mogę także wyświetlić boxplot pomiędzy tymi wartościami aby zobaczyć która opłata jest bliższa naszej wartości. Na czerwonej linii zaznaczyłem wartość 80.0 (czyli opłata która jest tu wypełniona)
ax = val.boxplot(column='Fare',by='Embarked');
ax.axhline(80,color='red')
Wartości są bardzo zbliżone, i wartość 80 jest bliska zarówno miediany dla ‘C’jak i dla ‘S‘. Ponieważ jednak port ‘C’ma ceny bardziej rozsztrzelone mogę podejrzewać że portem źródłowym jest ‘S’(Southampton).
full.set_value(62,'Embarked','S');
full.set_value(830,'Embarked','S');
full['_Embarked'] = pd.Categorical(full.Embarked).codes
Potrzeba także przeliczenia kolumny _Embarked żeby zawierała także kody.
Fare
Dla Fare postąpiłem podobnie, znalazłem najbardziej skorelowane zmienne i wyznaczyłem średnie wartości.
full[full['Fare'].isnull()]

full[cols].corr()['Fare'].abs().sort_values(ascending=False)
Fare 1.000000
Pclass 0.558629
_CabinType 0.547292
Survived 0.257307
_Embarked 0.238005
Parch 0.221539
_Sex 0.185523
Age 0.178740
SibSp 0.160238
_Title 0.028608
Name: Fare, dtype: float64
val = full[ (full['Pclass'] == 3)
& (full['Embarked'] == 'S')
& (full['Parch'] == 0)
& (full['Sex'] == 'male')
& (full['Age'] >40.0)
][['Age','Fare']];
val.groupby('Age').agg(['min','max','count','mean','median'])
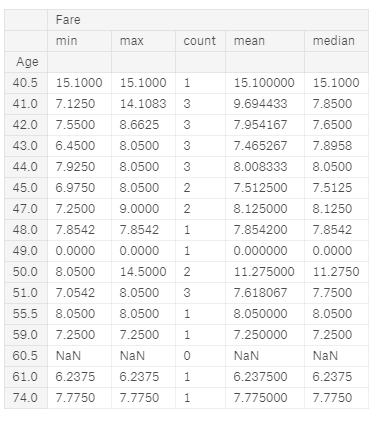
Można przypuszczać że osoby w tym wieku średnio płaciły między 6 a 7 funtów. Możemy w takim razie przyjąć średnią z tych dwóch wartości. Różnica między 6 a 7 jest zbyt mała aby mieć istotny wpływ w porównaniu do całego obszaru cen (od 0 do 512, gdzie średnia to 33 a mediana to 14)
full.at[1044,'Fare'] = (7.25 + 6.2375)/2; # we set average for this values
Age
Tutaj jest już spora ilość brakujących elementów, więc nie możemy jak poprzednio uzupełniać pojedyńcze wartości. Z pomocą idzie Scikit-learn który potrafi wartośći uzupełnić na podstawie średniej.
from sklearn.preprocessing import Imputer
imp = Imputer(missing_values='NaN', strategy='mean', axis=0)
full['_AgeImputer'] = imp.fit_transform(full[['Age']])
Cabin
Dla Cabin możemy użyć uzupełnienia przez najczęściej występujący element, ale nie chcemy używać elementu Cabin ponieważ jest za bardzo rozproszony. Dlatego użyjemy _CabinType. Najpierw wypełniamy go np.NANA
from sklearn.preprocessing import Imputer
full.loc[full['Cabin'].isnull(),'_CabinType'] = np.NAN
imp = Imputer(missing_values='NaN', strategy='most_frequent', axis=0)
full['_CabinType'] = imp.fit_transform(full[['_CabinType']])
full.at[1304,'_CabinType']
2.0
Puste wartości zostały wypełnione wartością 2.0.
full.isnull().sum()
Age 263
Cabin 1014
Embarked 0
Fare 0
Name 0
Parch 0
Pclass 0
Sex 0
SibSp 0
Survived 418
Ticket 0
_Sex 0
_Embarked 0
_CabinType 0
Title 0
_Title 0
_AgeImputer 0
AgeCategory 0
dtype: int64
Nie licząć Age (które nie będzie brało udziału), Cabin które nie wypełnialiśmy. Nie ma już pustych wartości
Python / Machine Learning
Teraz przyszedł czas na uczenie maszynowe i przepowiadanie wartości.
W tym wypadku trzeba podjąć 3 kroki:
- Znormalizować wszystkie kolumny biorące udział w liczeniu, w tym wypadku tylko kolumna Fare ponieważ kolumna Age została skategoryzowana.
- Wybrać najlepszy model na podstawie score wybrać model z najlepszym wynikiem.
- Przewidzieć wyniki i wysłać do sprawdzenia.
Normalizacja
Prawie wszystkie kolumny oprócz Fare są kolumnami kategorii. Fare warto zawsze przeskalować przed kalkulacjami tak żeby nie wpływał za bardzo na model.
from sklearn import preprocessing
full['_Fare'] = preprocessing.scale(full[['Fare']]) [:,0]
AgeCategory
- Warto także dodać AgeCategory który dzieli nam wiek na poszczególne kategorie. Ja podzieliłem go na 6 kategorii.
full['AgeCategory'] = pd.cut(full['_AgeLinear'],[0,9,18,30,40,50,100], labels=[9,18,30,40,50,100]) # Add column with range of Age
full['_AgeCategory'] = full['AgeCategory'].cat.codes # Add column with range of Age
Kolumny
Kolumny biorące udział w obliczeniach. Wszystkie kolumny są albo typu liczbowego albo kategorii. Powoduje to że mogą brać udział w klasyfikatorze.
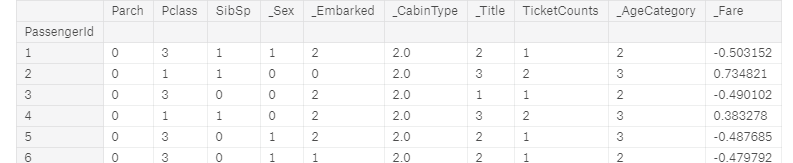
cols = ['Parch','Pclass','SibSp','_Sex','_Embarked','_CabinType','_Title','TicketCounts','_AgeCategory','_Fare']
full[cols]
ML - Import
Wszystkie klasyfikujące algorytmy mają podobny zestaw funkcji.
| Nazwa | Opis |
|---|---|
fit(X,Y) | Dopasuje klasyfikator do danaych |
score(X,Y) | Wyświetla jak bardzo dobrze poradził sobie klasyfikator |
predict(X) | Dla danego modelu przeprowadza obliczenia i przewiduje wyniik |
- Poniżej podałem większość klasyfikatorów dostępnych i na ich podstawie sprawdzimy które są najlepsze
from sklearn.linear_model import LogisticRegression, SGDClassifier
from sklearn.naive_bayes import MultinomialNB, BernoulliNB,GaussianNB
from sklearn.svm import SVC, LinearSVC, NuSVC
from sklearn import linear_model
from xgboost import XGBClassifier #conda install -c mndrake/xgboost (Windows 64)
classifiers = {
'Linear': linear_model.LogisticRegression(),
'SGDClassifier': SGDClassifier(),
'SVC': SVC(class_weight='balanced'),
'LinearSVC': LinearSVC(),
'NuSVC': NuSVC(),
'GaussianNB': GaussianNB(),
'BernoulliNB': BernoulliNB(),
'XGBoost': XGBClassifier()
# 'MultinomialNB': MultinomialNB()
}
Podział na Train i Test
- Do podziału na dane testowe i treningowe przydzia się funkcja train_test_split z sklearn. Najpierw pobieramy
X,Yktóre może zawierać tylko dane train dostępne od Kaggle:SURVdla naszych kolumn, następnie dzieliemy na dane treningowe i testowe.
from sklearn.model_selection import train_test_split
X = full[:SURV][cols] # ,'Parch','Embarked']]
Y = full[:SURV]['Survived']
X_train, X_test, y_train, y_test = train_test_split(X, Y, test_size=0.10)
Fit i Score
Teraz dla każdego klasyfikatora uczymy go clf.fit(X_train,y_train) a następnie obliczamy dokładność naszego modelu na danych testowych clf.score(X_test,y_test)
score = {}
for c in classifiers:
clf = classifiers[c]
clf.fit(X_train,y_train)
score[c] = clf.score(X_test,y_test)
#result[c] = clf.predict(Xp)
score
{'BernoulliNB': 0.8,
'GaussianNB': 0.7888888888888889,
'Linear': 0.7555555555555555,
'LinearSVC': 0.7444444444444445,
'NuSVC': 0.7777777777777778,
'SGDClassifier': 0.7888888888888889,
'SVC': 0.8111111111111111,
'XGBoost': 0.8111111111111111}
- Jak widać nie są to oszałamiające, ale wystarczą do końcowego kodu. Najlepiej poradził sobie XGboost i to jego wybierzemy jako model do przewidywań.
clf = classifiers['XGBoost']
Teraz pobieramy dane do predykcji Xp i wykorzystując clf.predict zapisujemy je do kolumny “Survived”. Zamiana jest na typ int , ponieważ Kaggle wymaga aby wynikiem była pojedyńcza liczba 1 i 0 bez przecinka
Xp = full[SURV:][cols]
result = pd.DataFrame({'PassengerID': full[SURV:].index })
result['Survived'] = clf.predict(Xp).T.astype(int)
Na koniec zapisujemy wynik do submission za pomocą to_csv
result[['PassengerID','Survived']].to_csv('submission.csv',index=False)
Result
Teraz możemy uruchomić cały kod i wysłać wynik do sprawdzenia. W tym celu:
- klikamy “Commit & Run”
- Przechodzimy do “Output”i klikamy “Submit to Competition”przy naszym pliku.
- Wynik 0.76 nie jest oszałamiający, ale na początek wystarczy. Jest wiele rzeczy które można poprawić w modelu i uzyskać lepszy wynik.
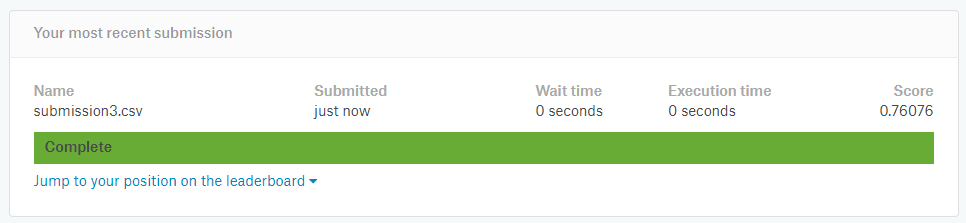
Python/ Końcowy kod
import numpy as np # linear algebra
import pandas as pd # data processing, CSV file I/O (e.g. pd.read_csv)
from subprocess import check_output
print(check_output(["ls", "../input"]).decode("utf8"))
# Load the data, we set that index_col is the first column, therefore there will be standard index start from 0 for each data.
train_df = pd.read_csv('../input/train.csv', header=0,index_col=0)
test_df = pd.read_csv('../input/test.csv', header=0,index_col=0)
full = pd.concat([train_df , test_df]) # concatenate two dataframes
# INFORMATION SECTION
full.info() # info about dataframe
full.head()
full[["Age","Pclass"]][5:30]
full[:10] # Pierwszych 10 elementów
full[-5:]
SURV = 891
full[SURV:SURV+10:2] # Like in regular Python you can get to the Item by Index
full[(full['Age'] > 5.0) & (full['Age'] < 7.0 ) ] #filter data by columns
full[(full['Cabin'].str.contains('B2',na=False)) ] #filter data by columns
full[full['Embarked'].isnull()]
# SUMMARY
full.isnull().sum() # Check with alues are empty
#Missing values in the plot
import missingno as msno
msno.matrix(full)
# GROUP
train_df.groupby(['Pclass','Sex'])['Survived'].sum() # grouping data
# DATA SECTION
full['_Sex'] = pd.Categorical(full.Sex).codes
full['_Embarked'] = pd.Categorical(full.Embarked).codes
full['_CabinType'] = pd.Categorical(full['Cabin'].astype(str).str[0]).codes
cols = ['Age','_Embarked','Fare','Parch','Pclass','_Sex','SibSp','Survived','_CabinType']
full[cols].corr()
pat = r",\s([^ .]+)\.?\s+"
full['Title'] = full['Name'].str.extract(pat,expand=True)[0]
full.groupby('Title')['Title'].count()
full.loc[full['Title'].isin(['Mlle','Ms','Lady']),'Title'] = 'Miss'
full.loc[full['Title'].isin(['Mme']),'Title'] = 'Mrs'
full.loc[full['Title'].isin(['Sir']),'Title'] = 'Mr'
full.loc[~full['Title'].isin(['Miss','Master','Mr','Mrs']),'Title'] = 'Other' # NOT IN
full['_Title'] = pd.Categorical(full.Title).codes
full.groupby('Title')['Title'].count()
full['TicketCounts'] = full.groupby(['Ticket'])['Ticket'].transform('count')
# WYKRESY
full['Age'].hist();
full[full['Survived']==1]['Age'].hist(bins=30)
import matplotlib
import matplotlib.pyplot as plt
plt_all = plt.hist(full['Age'],bins = 30, range = [0,100],label='all')
plt_survived =plt.hist(full[full['Survived']==1]['Age'], bins = 30, range = [0,100],label='Survived')
plt.legend()
plt.show()
## boxplot
full.boxplot(column='Age')
full.boxplot(column='Age',by='Pclass')
## dendrogram
import missingno as msno
msno.dendrogram(full)
cols = ['Age','_Embarked','Fare','Parch','Pclass','_Sex','SibSp','Survived','_CabinType']
corr = full[cols].corr()
## heatmap
import seaborn as sns
sns.heatmap(corr)
## wykres zbiorowy
for column in ['Pclass','Sex','SibSp','Parch','Embarked']:
fig, axes = plt.subplots(nrows=1, ncols=2)
(train_df
.groupby(column)['Survived']
.agg(['count','sum'])
).plot.bar(ax=axes[0])
(train_df
.groupby(column)['Survived']
.mean()
).plot.bar(ax=axes[1])
## ticket counts
(full
.groupby('TicketCounts')['Survived']
.agg(['count','sum'])
).plot.bar()
plt.show()
(full
.groupby('TicketCounts')['Survived']
.mean()
).plot.bar()
plt.show()
# Uzupełnianie pustych wartości
## NaN: Embarked
full[full['Embarked'].isnull()]
abs(full[cols].corr()['_Embarked']).sort_values(ascending=False)
val = full[ (full['Pclass'] == 1)
& (full['_CabinType'] == 1)
][['Fare','Embarked']];
val.groupby(['Embarked' ])['Fare'].agg(['count','min','max','mean','median'])
ax = val.boxplot(column='Fare',by='Embarked');
ax.axhline(80,color='red')
full.set_value(62,'Embarked','S');
full.set_value(830,'Embarked','S');
full['_Embarked'] = pd.Categorical(full.Embarked).codes
## NaN: Fare
full[full['Fare'].isnull()]
full[cols].corr()['Fare'].abs().sort_values(ascending=False)
val = full[ (full['Pclass'] == 3)
& (full['Embarked'] == 'S')
& (full['Parch'] == 0)
& (full['Sex'] == 'male')
& (full['Age'] >40.0)
][['Age','Fare']];
val.groupby('Age').agg(['min','max','count','mean','median'])
full.at[1044,'Fare'] = (7.25 + 6.2375)/2; # we set average for this values
## NaN: Age
from sklearn.preprocessing import Imputer
imp = Imputer(missing_values='NaN', strategy='mean', axis=0)
full['_AgeImputer'] = imp.fit_transform(full[['Age']])
## NaN: Cabin
from sklearn.preprocessing import Imputer
full.loc[full['Cabin'].isnull(),'_CabinType'] = np.NAN
imp = Imputer(missing_values='NaN', strategy='most_frequent', axis=0)
full['_CabinType'] = imp.fit_transform(full[['_CabinType']])
full.at[1304,'_CabinType']
## Verify null
full.isnull().sum()
# Process _Fare
from sklearn import preprocessing
full['_Fare'] = preprocessing.scale(full[['Fare']]) [:,0]
# Calculate AgeCategory
full['AgeCategory'] = pd.cut(full['_AgeImputer'],[0,9,18,30,40,50,100], labels=[9,18,30,40,50,100]) # Add column with range of Age
full['_AgeCategory'] = full['AgeCategory'].cat.codes # Add column with range of Age
# Select columns
cols = ['Parch','Pclass','SibSp','_Sex','_Embarked','_CabinType','_Title','TicketCounts','_AgeCategory','_Fare']
full[cols]
# Import models
from sklearn.linear_model import LogisticRegression, SGDClassifier
from sklearn.naive_bayes import MultinomialNB, BernoulliNB,GaussianNB
from sklearn.svm import SVC, LinearSVC, NuSVC
from xgboost import XGBClassifier #conda install -c mndrake/xgboost (Windows 64)
from sklearn import linear_model
classifiers = {
'Linear': linear_model.LogisticRegression(),
'SGDClassifier': SGDClassifier(),
'SVC': SVC(class_weight='balanced'),
'LinearSVC': LinearSVC(),
'NuSVC': NuSVC(),
'GaussianNB': GaussianNB(),
'BernoulliNB': BernoulliNB(),
'XGBoost': XGBClassifier()
# 'MultinomialNB': MultinomialNB()
}
# Podział na train i test
from sklearn.model_selection import train_test_split
X = full[:SURV][cols] # ,'Parch','Embarked']]
Y = full[:SURV]['Survived']
X_train, X_test, y_train, y_test = train_test_split(X, Y, test_size=0.10)
score = {}
for c in classifiers:
clf = classifiers[c]
clf.fit(X_train,y_train)
score[c] = clf.score(X_test,y_test)
#result[c] = clf.predict(Xp)
score
# Wybór najlepszego klasyfikatora
clf = classifiers['XGBoost']
# Obliczenie wyniku
Xp = full[SURV:][cols]
result = pd.DataFrame({'PassengerID': full[SURV:].index })
result['Survived'] = clf.predict(Xp).T.astype(int)
# Zapisanie wyniku
result[['PassengerID','Survived']].to_csv('submission.csv',index=False)
print('hurra, we find the result.')
Python/ Ulepszanie modelu
Wynik który uzyskaliśmy dla takiego zadania da się na pewno poprawić. na Kaggle nawet można zobaczyć wyniki wynoszące 1.0. Taki wynik może znaczyć że użytkownicy prawdopodobnie nie używali modelu a mają już zapisane wyniki które wcześniej przetestowali ponieważ prawie niemożliwością jest za pomocą modeli uzyskanie idealnego wyniku.
Na początku może zobaczymy na kod z którego usunąłem niepotrzebne elementy.
# This Python 3 environment comes with many helpful analytics libraries installed
# It is defined by the kaggle/python docker image: https://github.com/kaggle/docker-python
# For example, here's several helpful packages to load in
import numpy as np # linear algebra
import pandas as pd # data processing, CSV file I/O (e.g. pd.read_csv)
# Input data files are available in the "../input/" directory.
# For example, running this (by clicking run or pressing Shift+Enter) will list the files in the input directory
import os
print(os.listdir("../input"))
# Any results you write to the current directory are saved as output.
# Load the data, we set that index_col is the first column, therefore there will be standard index start from 0 for each data.
train_df = pd.read_csv('../input/train.csv', header=0,index_col=0)
test_df = pd.read_csv('../input/test.csv', header=0,index_col=0)
full = pd.concat([train_df , test_df]) # concatenate two dataframes
# DATA SECTION
full['_Sex'] = pd.Categorical(full.Sex).codes
full['_Embarked'] = pd.Categorical(full.Embarked).codes
full['_CabinType'] = pd.Categorical(full['Cabin'].astype(str).str[0]).codes
pat = r",\s([^ .]+)\.?\s+"
full['Title'] = full['Name'].str.extract(pat,expand=True)[0]
full.loc[full['Title'].isin(['Mlle','Ms','Lady']),'Title'] = 'Miss'
full.loc[full['Title'].isin(['Mme']),'Title'] = 'Mrs'
full.loc[full['Title'].isin(['Sir']),'Title'] = 'Mr'
full.loc[~full['Title'].isin(['Miss','Master','Mr','Mrs']),'Title'] = 'Other' # NOT IN
full['_Title'] = pd.Categorical(full.Title).codes
full['TicketCounts'] = full.groupby(['Ticket'])['Ticket'].transform('count')
# FILL N/A Values
full.at[1044,'Fare'] = (7.25 + 6.2375)/2; # we set average for this values
full.at[1304,'_CabinType']
## NaN: Age
from sklearn.preprocessing import Imputer
imp = Imputer(missing_values='NaN', strategy='mean', axis=0)
full['_AgeImputer'] = imp.fit_transform(full[['Age']])
## Verify null
# full.isnull().sum()
# Process _Fare
from sklearn import preprocessing
full['_Fare'] = preprocessing.scale(full[['Fare']]) [:,0]
# # Calculate AgeCategory
full['AgeCategory'] = pd.cut(full['_AgeImputer'],[0,9,18,30,40,50,100], labels=[9,18,30,40,50,100]) # Add column with range of Age
full['_AgeCategory'] = full['AgeCategory'].cat.codes # Add column with range of Age
# Select columns
cols = ['Parch','Pclass','SibSp','_Sex','_Embarked','_CabinType','_Title','TicketCounts','_AgeCategory','_Fare']
SURV = 891
X = full[:SURV][cols] # ,'Parch','Embarked']]
Y = full[:SURV]['Survived']
# Classifier
# conda install py-xgboost
from xgboost import XGBClassifier
clf = XGBClassifier()
clf.fit(X,Y)
# clf.score(X,Y) 0.8843995510662177
Xp = full[SURV:][cols]
result = pd.DataFrame({'PassengerID': full[SURV:].index })
result['Survived'] = clf.predict(Xp).T.astype(int)
result[['PassengerID','Survived']].to_csv('submission.csv',index=False)
print('hurra, we find the result.')
Automatyzacja wysyłania na kaggle:
LIMITY
Kaggle ma limity na wysyłanie odpowiedzi do 10 w ciągu dnia. Dlatego radze mieć to na uwadze.
Wysyłanie wyników na serwer przez stronę Kaggle jest uciążliwe, dlatego istnieje automat do wysyłania naszych wyników.
Wystarczy zainstalować kaggle:
pip install kaggle
Wrzucić nasz API-Key do konfiguracji użytkownika:
https://github.com/Kaggle/kaggle-api
I można wygodnie wysyłać z konsoli nasze wyniki
kaggle competitions submit -c titanic -f submission.csv -m "Message"
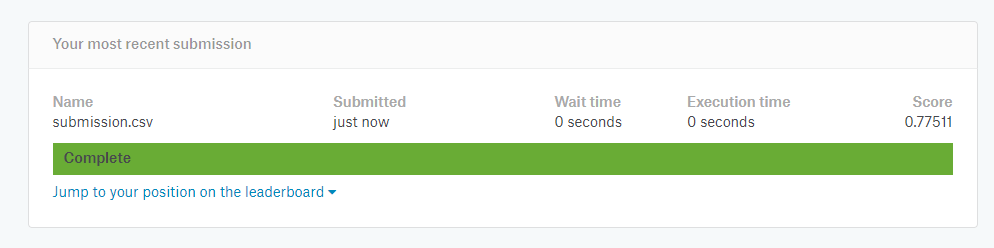
Na stronię https://www.kaggle.com/c/titanic/submissions mamy możliwość przejrzenia naszych wyników.
Na początku mamy wynik 0.76555:
Od tej chwile będe używał lokalnego kodu i sprawadzał wyniki bezpośrednio.
Co można poprawić w takim kodzie?
- Imputer wpisuje jedną wartość dla wszystkich identyczną, fajnie by było gdyby można było wyznaczyć wiek na podstawie innych kolumn.
- Można sparametryzować XGBoost dla lepszego dopasowania
- Można użyć GridSearchCV do automatycznego sparametryzowania
Poprawianie pustych wartości dla Age
Do poprawienia tej kolumny użyłem Linnear Regression. Najpierw wyznaczyłem parametry które najbardziej wpływają na wiek przez:
full[['Age','_Embarked','Fare','Parch','Pclass','_Sex','SibSp','Survived','_CabinType','_Title']].corr()['Age'].abs().sort_values(ascending=False)
Potem uzyskałem wynik 0.77033, natomiast jeśli wyznaczyłem wiek na podstawie kolumn:
cols = ['Pclass','_CabinType','SibSp','Fare','Parch']
ageData = full[full['Age'].notnull()]
emptyData = full[full['Age'].isnull()]
Y = ageData['Age']
X = ageData[cols] # ,'Parch','Embarked']]
# Create linear regression object
from sklearn import linear_model
regr = linear_model.LinearRegression()
regr.fit(X,Y)
X = emptyData[cols]
Y = regr.predict(X)
# First we need to set index before
pred = pd.concat([pd.Series(Y,emptyData.index),ageData['Age']]).sort_index()
full['_AgeLinear'] = pred
# Calculate AgeCategory
full['AgeCategory'] = pd.cut(full['_AgeLinear'],[0,9,18,30,40,50,100], labels=[9,18,30,40,50,100]) # Add column with range of Age
full['_AgeCategory'] = full['AgeCategory'].cat.codes # Add column with range of Age
Wynik podskoczył do: 0.776
Poprawianie pustych wartości dla _CabinType
Tutaj też mogłem zastosować linear regression albo jakieś DecicionTree ponieważ chciałbym tylko sklasyfikować te elementy. W tym wypadku skasowałem Imputer dla _CabinType (LinearRegression może sobie poradzić z pustymi wartościami) a następnie przeniosłem kod dalej:
from sklearn.tree import DecisionTreeClassifier
clas = DecisionTreeClassifier(criterion = "gini", random_state = 100,
max_depth=3, min_samples_leaf=5)
cols = ['Pclass','Fare','_AgeLinear','_Embarked','_Title', '_Sex']
data = full[full['Cabin'].notnull()]
emptyData = full[full['Cabin'].isnull()]
X = data[cols]
Y = data['_CabinType']
clas.fit(X,Y)
X = emptyData[cols]
Y = clas.predict(X)
# First we need to set index before
pred = pd.concat([pd.Series(Y,emptyData.index), data['_CabinType']]).sort_index()
full['CabinType'] = pred
Wynik podskoczył do:
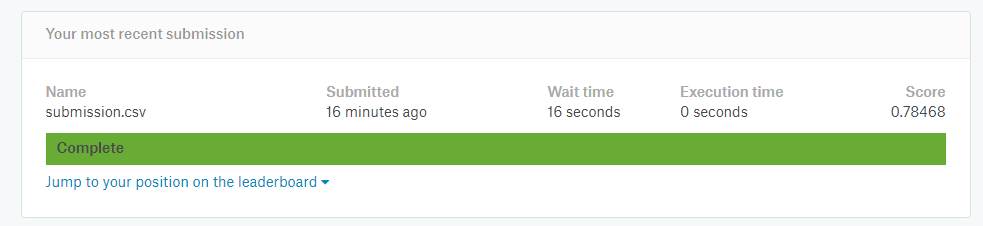
Pełny kod:
import numpy as np # linear algebra
import pandas as pd # data processing, CSV file I/O (e.g. pd.read_csv)
import os
# print(os.listdir("../input"))
train_df = pd.read_csv('../input/train.csv', header=0,index_col=0)
test_df = pd.read_csv('../input/test.csv', header=0,index_col=0)
full = pd.concat([train_df , test_df]) # concatenate two dataframes
# DATA SECTION
full['_Sex'] = pd.Categorical(full.Sex).codes
full['_Embarked'] = pd.Categorical(full.Embarked).codes
full['_CabinType'] = pd.Categorical(full['Cabin'].astype(str).str[0]).codes
pat = r",\s([^ .]+)\.?\s+"
full['Title'] = full['Name'].str.extract(pat,expand=True)[0]
full.loc[full['Title'].isin(['Mlle','Ms','Lady']),'Title'] = 'Miss'
full.loc[full['Title'].isin(['Mme']),'Title'] = 'Mrs'
full.loc[full['Title'].isin(['Sir']),'Title'] = 'Mr'
full.loc[~full['Title'].isin(['Miss','Master','Mr','Mrs']),'Title'] = 'Other' # NOT IN
full['_Title'] = pd.Categorical(full.Title).codes
full['TicketCounts'] = full.groupby(['Ticket'])['Ticket'].transform('count')
# FILL N/A Values
full.at[1044,'Fare'] = (7.25 + 6.2375)/2; # we set average for this values
## NaN: Age
# fill using LinearRegression
# full[['Age','_Embarked','Fare','Parch','Pclass','_Sex','SibSp','Survived','_CabinType','_Title']].corr()['Age'].abs().sort_values(ascending=False)
cols = ['Pclass','_CabinType','SibSp','Fare','Parch']
ageData = full[full['Age'].notnull()]
emptyData = full[full['Age'].isnull()]
Y = ageData['Age']
X = ageData[cols] # ,'Parch','Embarked']]
# Create linear regression object
from sklearn import linear_model
regr = linear_model.LinearRegression()
regr.fit(X,Y)
X = emptyData[cols]
Y = regr.predict(X)
# First we need to set index before
pred = pd.concat([pd.Series(Y,emptyData.index),ageData['Age']]).sort_index()
full['_AgeLinear'] = pred
# Calculate AgeCategory
full['AgeCategory'] = pd.cut(full['_AgeLinear'],[0,9,18,30,40,50,100], labels=[9,18,30,40,50,100]) # Add column with range of Age
full['_AgeCategory'] = full['AgeCategory'].cat.codes # Add column with range of Age
# Cabin Type
# full[['_AgeLinear','_Embarked','Fare','Parch','Pclass','_Sex','SibSp','Survived','_CabinType','_Title']].corr()['_CabinType'].abs().sort_values(ascending=False)
from sklearn.tree import DecisionTreeClassifier
clas = DecisionTreeClassifier(criterion = "gini", random_state = 100,
max_depth=3, min_samples_leaf=5)
cols = ['Pclass','Fare','_AgeLinear','_Embarked','_Title', '_Sex']
data = full[full['Cabin'].notnull()]
emptyData = full[full['Cabin'].isnull()]
X = data[cols]
Y = data['_CabinType']
clas.fit(X,Y)
X = emptyData[cols]
Y = clas.predict(X)
# First we need to set index before
pred = pd.concat([pd.Series(Y,emptyData.index), data['_CabinType']]).sort_index()
full['CabinType'] = pred
# Process Normalization Fare
from sklearn import preprocessing
full['_Fare'] = preprocessing.scale(full[['Fare']]) [:,0]
# conda install py-xgboost
# Select columns
cols = ['Parch','Pclass','SibSp','_Sex','_Embarked','_CabinType','_Title','TicketCounts','_AgeCategory','_Fare']
SURV = 891
X = full[:SURV][cols] # ,'Parch','Embarked']]
Y = full[:SURV]['Survived']
# Classifier
from xgboost import XGBClassifier
clf = XGBClassifier()
clf.fit(X,Y)
print(clf.score(X,Y)) # 0.8843995510662177
Xp = full[SURV:][cols]
result = pd.DataFrame({'PassengerID': full[SURV:].index })
result['Survived'] = clf.predict(Xp).T.astype(int)
result[['PassengerID','Survived']].to_csv('submission.csv',index=False)
print('hurra, we find the result.')
Poprawianie XGBoost
XGBoost jest algorytmem opartym na drzewie decyzyjnym. Jak pewnie zauważyliście dla algorytmu XGBoost nie podałem żadnych parametrów. W takim razie możemy zmienić kilka z nich, możliwe że się poprawi wynik.
Całą listę parametrów do poprawiania można znaleść na stronie
a opis niektórych z nich:
Najważniejszy z nich to learning_rate, pozwala określić jak bardzo zmieniamy wagi w sieci przy naszej funkcji błędu, za niski spowoduje długie uczenie sieci, za duże spowoduje że sieć będzie błąd będzie zbyt duży. Reszta parametrów dałem jako przykład.
from xgboost import XGBClassifier
clf = XGBClassifier(
learning_rate =0.1,
n_estimators=10000,
max_depth=5,
min_child_weight=1,
gamma=0,
subsample=0.8,
colsample_bytree=0.8,
objective= 'binary:logistic',
nthread=4,
scale_pos_weight=1,
seed=27)
Po tym wszystkim zobaczyłem jaki jest score dla mojej sieci lokalnie (dla danych treningowych), i wyszło 0.955, ale po przejsłaniu wyniku na serwer okazało się że wynik jaki dostaje to marne 0.72 😞
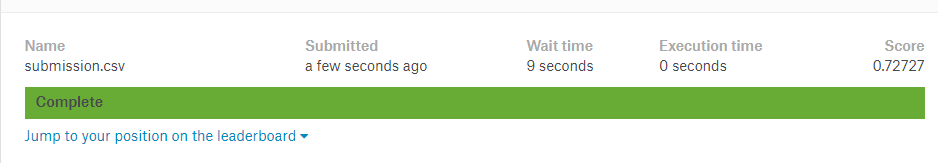
Po tym wszystkim zobaczyłem jaki jest score dla mojej sieci lokalnie (dla danych treningowych), i wyszło 0.955, ale po przejsłaniu wyniku na serwer okazało się że wynik jaki dostaje to marne 0.72 😞

Co spowodowało taki spadek, a mianowicie przeuczenie modelu. XGBoost uzyskał tak dobry wynik dla danych treningowych że model nie poradził sobie z danymi testowymi tak dobrze, jak gdy nie dopasowała się aż tak dobrze poprzednio (miało odpowiednio score: 0.884 dla treningowych i 0.78 dla testu). Co obrazuje poniższy obrazek i przykład wzięty z uczenia sieci neuronowej.
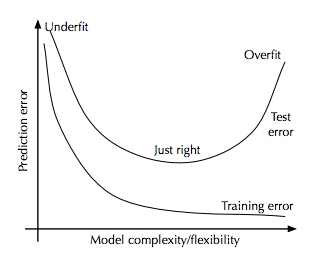
źródło: https://www.quora.com/Is-it-necessary-to-get-rid-of-useless-features-in-the-classification-task-in-XGBoost
źródło: http://www.grandjean-bpa.com/UL-nnfit/english/man/nnfit2_man.html
Jak widać ten model idealnie dopasował wartości ale czy dla wartości które nie zna jest aż tak dobre. Okazuje się że nie.
XGBoost - early_stopping_rounds
Do powstrzymania przez GridSearchCV d przeuczeniem można użyć early_stopping_rounds które pozwala zatrzymać uczenie jeśli model nie poprawia się przez dłuższy czas. Więcej szczegułów wyczytałem na:
https://machinelearningmastery.com/avoid-overfitting-by-early-stopping-with-xgboost-in-python/
# Classifier
from xgboost import XGBClassifier
clf = XGBClassifier(
learning_rate =0.01,
n_estimators=1000,
max_depth=5,
min_child_weight=1,
gamma=0,
subsample=0.8,
colsample_bytree=0.8,
objective= 'binary:logistic',
nthread=4,
scale_pos_weight=1,
seed=27,
eval_metric="error", verbose=True
)
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(X, Y, test_size=0.33)
eval_set = [(X_test, y_test)]
clf.fit(X,Y,early_stopping_rounds=10, eval_metric="logloss", eval_set=eval_set, verbose=True)
print(clf.score(X,Y)) # 0.8843995510662177
Xp = full[SURV:][cols]
result = pd.DataFrame({'PassengerID': full[SURV:].index })
result['Survived'] = clf.predict(Xp).T.astype(int)
Spowodowało to polepszenie modelu do 0.75119 .
XGBoost - GridSearchCV
GridSearchCV powoduje szukanie najlepszych parametrów dla XGBoost. Za jego pomocą zamast samemu zgadywać parametry można pozwolić algorytmowi na odgadnięcie najlepszych. My tylko podajemy w parametrach zakresy jakie powinny być.
param_test1 = {
'max_depth':range(3,10,2),
'min_child_weight':range(1,6,2)
}
from sklearn.model_selection import GridSearchCV
gsearch1 = GridSearchCV(estimator = clf,
param_grid = param_test1, scoring='roc_auc',n_jobs=4,iid=False, cv=5)
gsearch1.fit(X,Y)
gsearch1.score(X,Y)
Xp = full[SURV:][cols]
result = pd.DataFrame({'PassengerID': full[SURV:].index })
result['Survived'] = gsearch1.predict(Xp).T.astype(int)
result[['PassengerID','Survived']].to_csv('submission.csv',index=False)
print('hurra, we find the result.')
I wynik jaki uzyskałem to także 0.77511 :happy:
Jak widać ciężko jest uzyskać powyżej 0.8. Najbardziej prawdopodobne jest to że nie wyciągnąłem jeszcze innych informacji jak np: wielkość rodziny (które widziałem w innych przykładach). Radze pobawić się parametrami zobaczyć wyniki i posprawdzać co można jeszcze polepszyć.
R/ Wstęp
Zaczynając prace z Kaggle mamy tak naprawdę 3 możliwości:
- Możemy ściągnąć dane train i test i działać na nim lokalnie
- Możemy utworzyć Kernel i wysłać skrypt który rozwiąże zadanie
- Możemy utworzyć Kernel i za pomocą edytora “Jupyter Notebooks”nie tylko wykonać wszystkie operacje ale także dodać w wygodnym edytorze MarkDown własne przypisy czy wykresy.
Ja wybrałem Markdown bo można na bieżąco kontrolować co się dzieje. Jeśli chcesz utworzyć nowy kernel na stronie https://www.kaggle.com/c/titanic wybieramy Kernels -> New Kernel -> Notebook u góry po prawej możemy zmienić język z Python (który jest domyślny) na R.
R/ Podstawowe operacje
Podstawowe funkcje można znaleść w:
https://www.rstudio.com/wp-content/uploads/2016/10/r-cheat-sheet-3.pdf
https://s3.amazonaws.com/assets.datacamp.com/blog_assets/Tidyverse+Cheat+Sheet.pdf
https://www.rstudio.com/wp-content/uploads/2015/02/data-wrangling-cheatsheet.pdf
Biblioteki
Najpierw importujemy biblioteki
library(tidyverse) # metapackage with lots of helpful functions
list.files(path = "../input")
- tidyverse - kolekcja paczek do data-science, zawiera w sobie już wiele potrzebnych paczek
W R podobnie jak w python podstawowym obiektem na którym operujemy jest data.frame (w książce Biecka “Przewodnik po pakiecie R” nazywane także jako“ramki danych”) który jest 2 wymiarową tablicą z kolumnami i wierszami. W przypadku większych danych jest jeszcze struktura data.table ale dla 1309 tysiąca wierszy wystarczy zwykły data.frame.
read_csv()
train <- read_csv('../input/train.csv')
test <- read_csv('../input/test.csv')
import danych, funkcja już wie że pierwszy wiersz opisuje kolumny, nie trzeba to zaznaczać w metodzie.
rbind()
Połączenie wierszy danych, tak żeby móc zobaczyć pełną informację o danych. Nie można łączyć danych o różnych ilościach kolumn dlatego do kolumny test$Survived dodajemy pustą informację.
test$Survived <- NA
full <- rbind(train, test)
str()
Podaje podstawe informacje o danych, jak typ - przykłady wartości.
str(full)
Classes ‘tbl_df’, ‘tbl’ and 'data.frame': 1309 obs. of 12 variables:
$ PassengerId: num 1 2 3 4 5 6 7 8 9 10 ...
$ Survived : num 0 1 1 1 0 0 0 0 1 1 ...
$ Pclass : num 3 1 3 1 3 3 1 3 3 2 ...
$ Name : chr "Braund, Mr. Owen Harris" "Cumings, Mrs. John Bradley (Florence Briggs Thayer)" "Heikkinen, Miss. Laina" "Futrelle, Mrs. Jacques Heath (Lily May Peel)" ...
$ Sex : chr "male" "female" "female" "female" ...
$ Age : num 22 38 26 35 35 NA 54 2 27 14 ...
$ SibSp : num 1 1 0 1 0 0 0 3 0 1 ...
$ Parch : num 0 0 0 0 0 0 0 1 2 0 ...
$ Ticket : chr "A/5 21171" "PC 17599" "STON/O2. 3101282" "113803" ...
$ Fare : num 7.25 71.28 7.92 53.1 8.05 ...
$ Cabin : chr NA "C85" NA "C123" ...
$ Embarked : chr "S" "C" "S" "S" ...
- attr(*, "spec")=
.. cols(
.. PassengerId = col_double(),
.. Survived = col_double(),
.. Pclass = col_double(),
.. Name = col_character(),
.. Sex = col_character(),
.. Age = col_double(),
.. SibSp = col_double(),
.. Parch = col_double(),
.. Ticket = col_character(),
.. Fare = col_double(),
.. Cabin = col_character(),
.. Embarked = col_character()
.. )
dim()
Zwraca rozmiar data.frame (wiersze i kolumny )
dim(full)
nrow(full) #liczba kolumn
ncol(full) #liczba wierszy
1309 12
head()
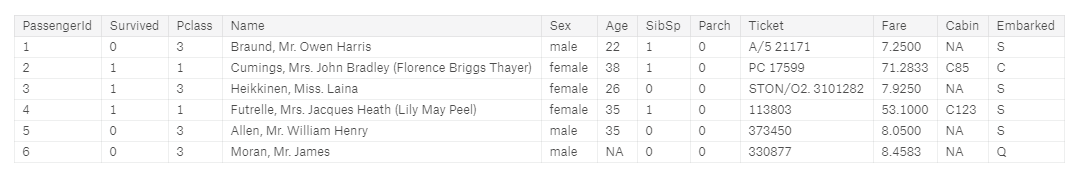
Zwraca kilka pierwszych wierszy do rozeznania się.
head(full)
summary()
summary(full)
Zwraca podstawowe informacje o kolumnach - jak liczba pustych wartości, wartość minimalna, maksymalna, mediana itp… Już widać że kilka kolumn ma puste wartości które to trzeba będzie uzupełnić póżniej. (NA’s)
PassengerId Survived Pclass Name
Min. : 1 Min. :0.0000 Min. :1.000 Length:1309
1st Qu.: 328 1st Qu.:0.0000 1st Qu.:2.000 Class :character
Median : 655 Median :0.0000 Median :3.000 Mode :character
Mean : 655 Mean :0.3838 Mean :2.295
3rd Qu.: 982 3rd Qu.:1.0000 3rd Qu.:3.000
Max. :1309 Max. :1.0000 Max. :3.000
NAs :418
Sex Age SibSp Parch
Length:1309 Min. : 0.17 Min. :0.0000 Min. :0.000
Class :character 1st Qu.:21.00 1st Qu.:0.0000 1st Qu.:0.000
Mode :character Median :28.00 Median :0.0000 Median :0.000
Mean :29.88 Mean :0.4989 Mean :0.385
3rd Qu.:39.00 3rd Qu.:1.0000 3rd Qu.:0.000
Max. :80.00 Max. :8.0000 Max. :9.000
NAs :263
Ticket Fare Cabin Embarked
Length:1309 Min. : 0.000 Length:1309 Length:1309
Class :character 1st Qu.: 7.896 Class :character Class :character
Mode :character Median : 14.454 Mode :character Mode :character
Mean : 33.295
3rd Qu.: 31.275
Max. :512.329
NAs :1
Wybieranie kolumn
full[,c("Survived","Pclass")]
- Lub przez select z dplyr
library(dplyr)
select(full,Survived,Pclass)
Filtrowanie
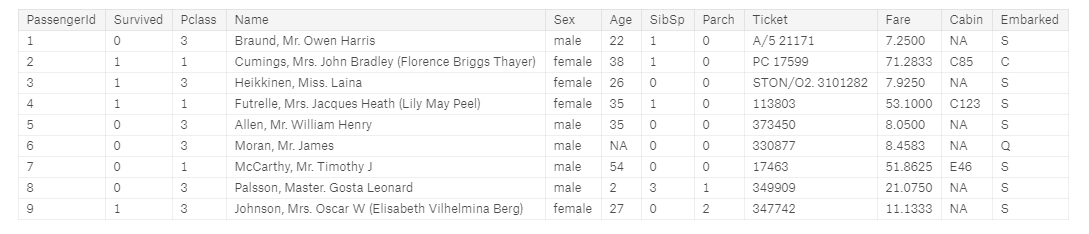
- Dane można zaznaczać przez indeksy
full[1:9,]
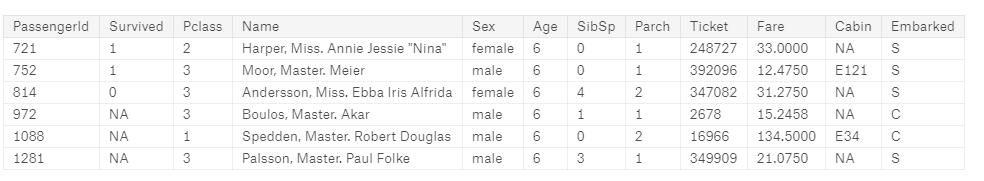
- Do bardziej zaawansowango filtrowania po wartościach używa się biblioteki dplyr. Można używać złączeń typu AND “&”OR “|” NOT “!”
library(dplyr)
filter(full, Age > 5.0 & Age < 7.0 )
- Dodatkowo można to kaskadować na kolejne wyrażenia z dplyr. W tym wypadku nie trzeba za każdym razem zaznaczać danych (full)
full %>%
filter(Age > 5.0 & Age < 7.0 ) %>%
select(Survived,Pclass)
- sprawdzanie pustych wartości
full %>%
filter( is.na(Fare) )
- Filtrowanie po tekście używająć stringr
https://cran.r-project.org/web/packages/stringr/vignettes/stringr.html
library(stringr)
full %>%
filter( str_detect(Cabin, 'B2') )
Grupowanie
- Do sumowania i groupowania przydaje się dplyr, tutaj wymagane jest jeszcze usunięcie pustych wartości survived ponieważ w przeciwnym wypadku pojawi się N/A w wyniku.
full %>%
drop_na(Survived) %>%
group_by(Pclass,Sex) %>%
summarise(Survived = sum(Survived))
- Można też nie używać drop_na a opcji na.rm=T
full %>%
group_by(Pclass,Sex) %>%
summarise(Survived = sum(Survived, na.rm = T))
R/ Wykresy i Dane
Tak naprawdę pod tym pojęciem chciałem napisać w jaki sposób można dodać kolumny zawierające dodatkowe informacje które kryją się w surowych danych ponieważ może się zdarzyć że:
- Jakieś informacje znajdują się w tekście a ponieważ znajdują się tam także inne informacje to algorytm nie wykryje że dana zmienna ma istotny wpływ
- Niektóre dane są zbyt rozdrobnione (np: wiek) przez co zaburzają obraz całego modelu. Przecięz podczas próby uratowania się nikt nie pytał dokładnie o wiek, więc można podzielić je na mniejsze grupy typu małe dzieci, dorośli itp..
- Część danych jest w postaci tekstowej, i trzeba je skategoryzować.
R/ Dane
Kategoryzacja
- W przypadku R kategoryzacja polega na przerobieniu na factor za pomocą as.factor a następnie na as.numeric
full$Sex2 <- as.numeric(as.factor(full$Sex))
full$Embarked2 <- as.numeric(as.factor(full$Embarked))
Kategoryzacja kolumny Cabin
full$Cabin2 <-as.numeric(as.factor(substring(full$Cabin, 0, 1) ))
Korelacja
- Korelacje można wwykonać przez funkcję
cols <- c('Age','Sex2','Embarked2','Fare','Parch','Pclass','SibSp','Survived','Cabin2')
cor( full[,cols], use="complete.obs")
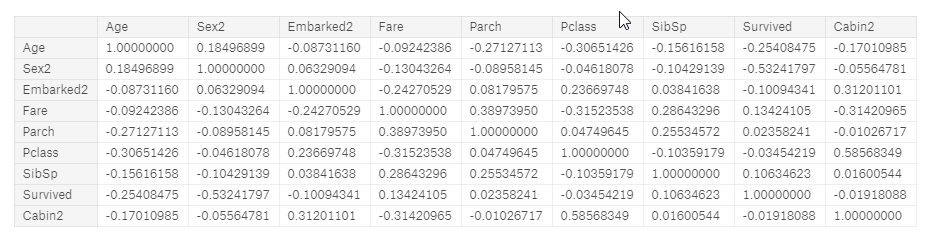
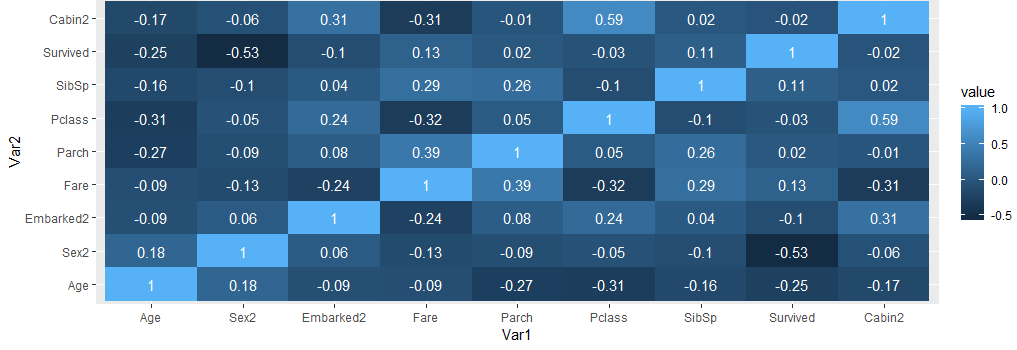
Widać największą korelację przy kolumnie Sex2, Age, Fare. To pomoże przy określaniu kolumn dostępnych do algorytmu.
Tytuł - Pattern
- Warto sobie wyciągnąć tytuł z imienia i nazwiska czyli kolumny Name. Do tego przydaje się regular expression
- Tytuł można wyłuskać przez str_match z biblioteki
library(stringr)
full$Title <- str_match(full$Name, ",\\s([^ .]+)\\.?\\s+")[,2]
full %>%
group_by(Title) %>%
summarise(cnt = n()) %>%
arrange(desc(cnt))
Title cnt
<chr> <int>
1 Mr 757
2 Miss 260
3 Mrs 197
4 Master 61
5 Dr 8
6 Rev 8
7 Col 4
8 Major 2
9 Mlle 2
10 Ms 2
11 Capt 1
12 Don 1
13 Dona 1
14 Jonkheer 1
15 Lady 1
16 Mme 1
17 Sir 1
18 the 1
- Część danych nie jest potrzebna, dlatego najlepiej zgrupować je sobie w kilka kategori do lepszego predykcji
full$Title2 <- full$Title
full$Title2[ full$Title %in% c('Mlle','Ms','Lady')] <- 'Miss'
full$Title2[ full$Title %in% c('Mme')] <- 'Mrs'
full$Title2[ full$Title %in% c('Sir')] <- 'Mr'
full$Title2[ ! full$Title %in% c('Miss','Master','Mr','Mrs')] <- 'Other'
full$TitleN <- as.numeric(as.factor(full$Title2))
full %>%
group_by(Title2) %>%
summarise(cnt = n()) %>%
arrange(desc(cnt))
Title2 cnt
<chr> <int>
1 Mr 757
2 Miss 260
3 Mrs 197
4 Master 61
5 Other 34
TicketCounts
- Wyciągnałem tak też nową kolumnę TicketCounts które oznacza ile osób ma ten sam numer biletu.
full <- full %>% group_by(Ticket) %>% mutate(TicketCount = n()) %>% ungroup()
R/ Wykresy
Histogram
histogram można wyświetlić przez prostą funkcję hist
hist(full$Age)
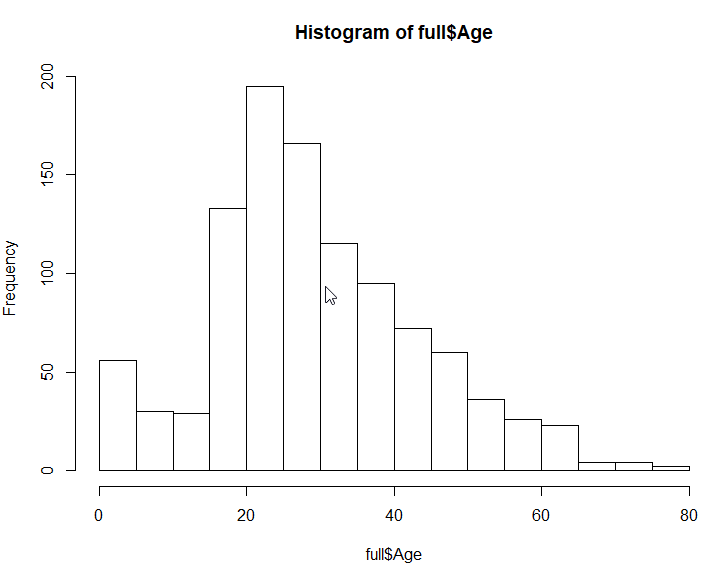
Ilość podziałów można ustawić przez breaks
hist(full$Age, breaks=60)
Histogram można takżę narysować przez funkcję ggplot.
ggplot(data=full, aes(full$Age)) + geom_histogram()
ggplot(data=full, aes(full$Age)) + geom_histogram(bins=60)
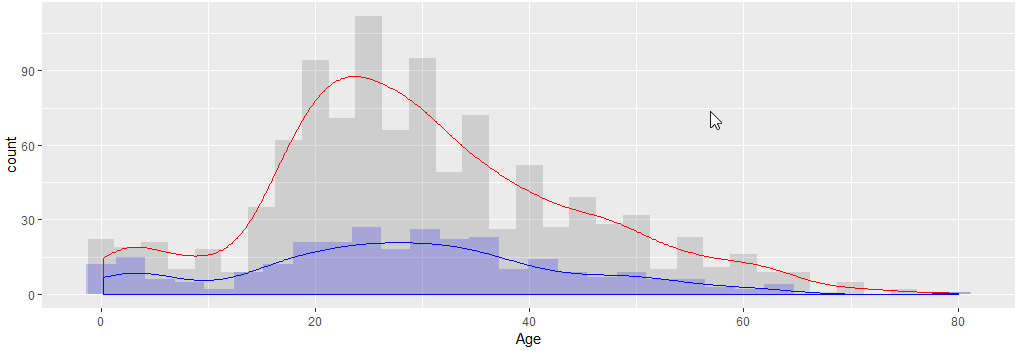
Dzięki ggplot można nawet nałożyć kilka wykresów (jak histogram)
ggplot(data=full, aes(x=Age)) +
geom_histogram(binwidth = 2.5, alpha = 0.2) +
geom_histogram(data = subset(full,Survived == 1), fill = "blue", alpha = 0.2) +
geom_density(aes(y=2.5 * ..count..),color="red",na.rm = T) +
geom_density(data = subset(full,Survived == 1), aes(y=2.5 * ..count..),color="blue",na.rm = T)
- geom_denisty - powoduje narysowanie szacowanej linii gęstości. Ponieważ wynik jest od 0 do 1, trzeba albo histogram zmniejszyć albo geom_density powiększyć do histogramu (
aes=(y=2.5* ..count..))
Boxplot
Zawiera on dużo informacji więc lepiej doczytać co oznaczają poszczególne wartości
https://www.wellbeingatschool.org.nz/information-sheet/understanding-and-interpreting-box-plots
ggplot(data=full, aes(Survived, Age, group = Survived)) + geom_boxplot()
Co można wywnionskować
- Mediana przeżycia jest równa co do wieku
- Zwykle młodsi przeżywają
ggplot(data=full, aes(Pclass, Age, group = Pclass)) + geom_boxplot()
Co można wywnioskować że
- Osoby z 3 klasy są najmłodsze i rozrzut jest najmniejszy ze wszystkich.
- 50% osób w pierwszej klasie znajduje się między 30 a 50 rokiem życia
Heatmap / Correlation Map
Prosty wykres korelacji danych można zrobić przez funkcję heatmap
cols <- c('Age','Sex2','Embarked2','Fare','Parch','Pclass','SibSp','Survived','Cabin2')
corr <- cor( full[,cols], use="complete.obs")
heatmap(corr,
na.rm = T, # same data set for cell labels
)
Bardziej zaawansowany heatmap oferuje ggplot, trzeba tylko wynik korelacji (który jest typu matrix) zamienić na data.frame. Można to zrobić przez funkcje melt
library(reshape2)
corr_df <- melt(corr, na.rm = TRUE)
ggplot(corr_df, aes(x=Var1,y=Var2, fill=value)) +
geom_tile() +
geom_text(aes(Var2, Var1, label = round(value,2)), color = "white", size = 4)
Performance Analytics
Dość ciekawym wykresem jest Performance Analytics. Pozwala na jedynm wykresie pokazać histogram, zależności między zmiennymi i jak zmieniają się dane w zależności od zmiennych.
cols <- c('Age','Sex2','Embarked2','Fare','Parch','Pclass','SibSp','Survived','Cabin2','TitleN')
# install.packages("PerformanceAnalytics")
library("PerformanceAnalytics")
chart.Correlation(full[,cols], histogram=TRUE, pch=19, font.size = 15)
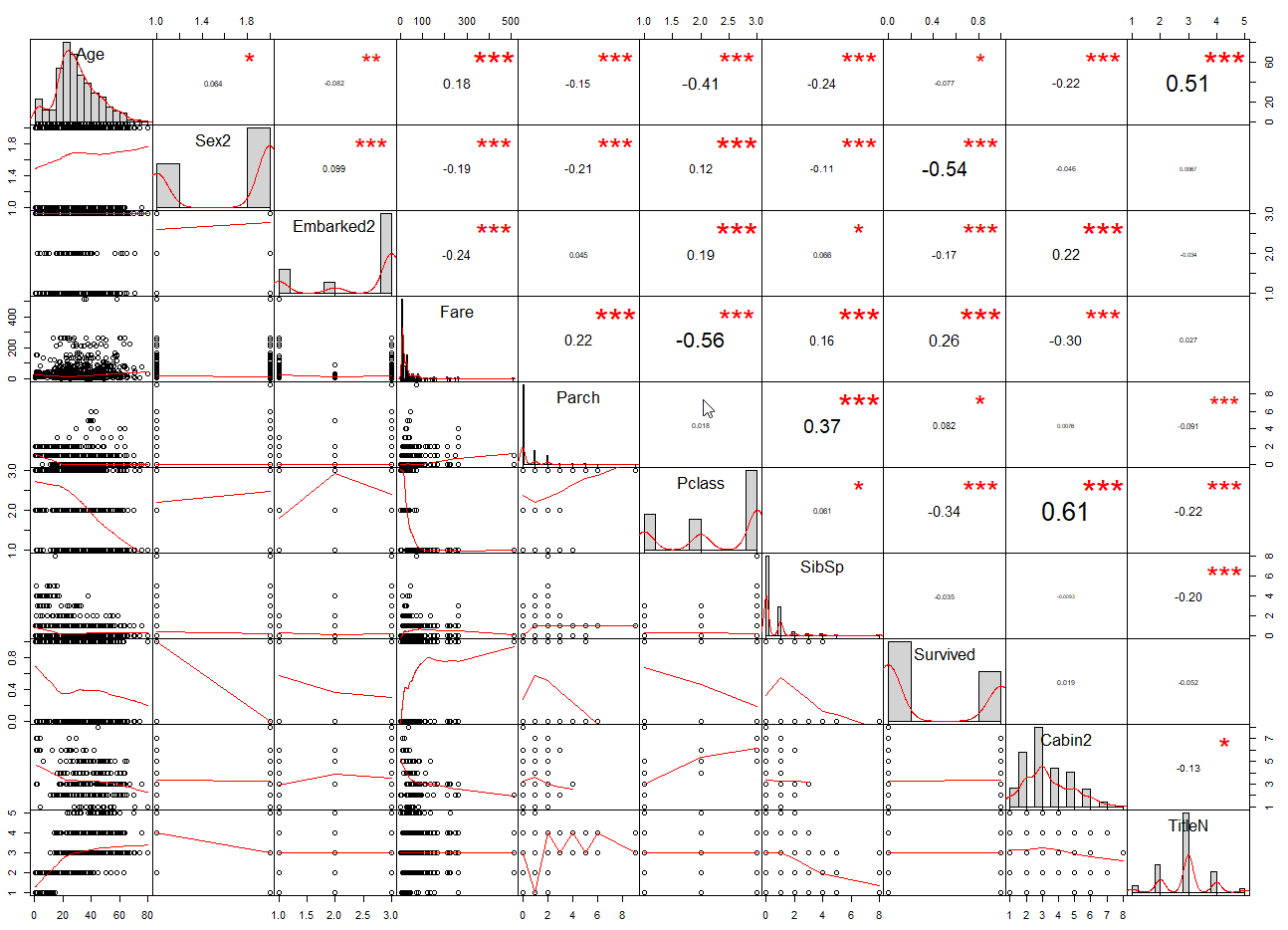
- Po przekątnej mamy histogram, po lewej na dole wykresy punktowe i szacowany wykres zależności między punktami.
- Np: wiersz Survived z kolumną Age widzimy żę wraz ze wzrostem wieku przeżywalność spada, odwrotnie jak w przypadku opłaty za bilet Fare, wraz ze wzrostem przeżywalność rośnie
- Na górze mamy corelację między zmiennymi.
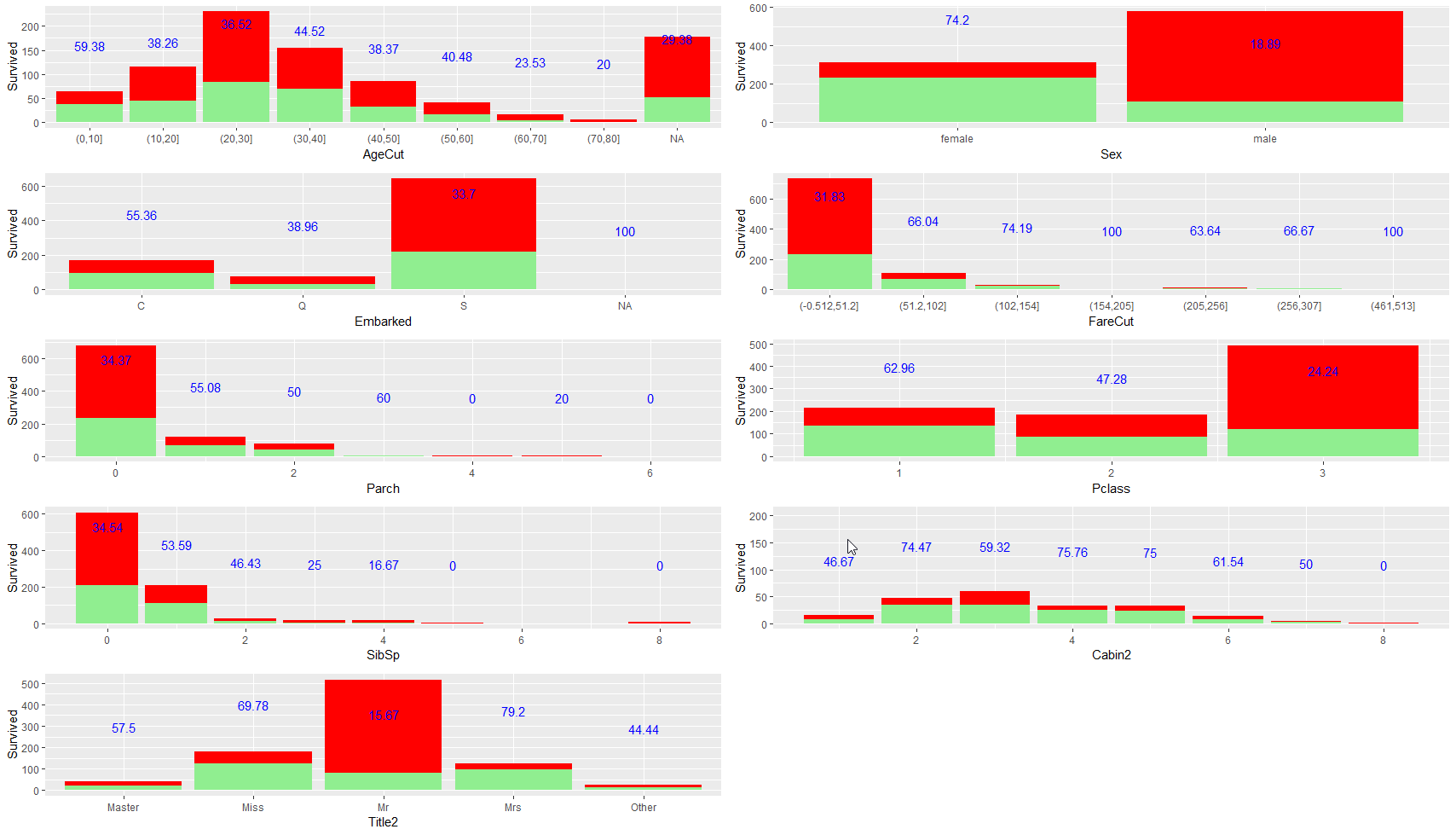
Grupowe analizy
Można także zgrupować wykresy w przeliczeniu na przeżywalność i umieścić je na jednym wykresie
W tym wypadku można użyć funkcji grid.arrange z gridExtra() a przedtem dla każdego wygenerować wykres który dodajemy do listy vector[[col]] <-plot
Ponieważ Age i Fare są wartościami ciągłymi dobrze jest je podzielić na obszary przez funkcje cut
library(ggplot2)
require(gridExtra)
full$AgeCut <- cut(full$Age,breaks = seq(0, 100, by = 10))
full$FareCut <- cut(full$Fare,10)
cols <- c('AgeCut','Sex','Embarked','FareCut','Parch','Pclass','SibSp', 'Cabin2','Title2')
vector <- list()
get_plot <- function (data, col) {
data_group <- data[,c(col,"Survived")] %>%
select(x = col, "Survived") %>%
drop_na(Survived) %>%
group_by(x) %>%
summarise(Total = n(),Survived = sum(Survived,na.rm = T), ratio = sum(Survived,na.rm = T)/n())
plot <- ggplot(data_group ,aes(x, Survived, label='A')) +
geom_bar(aes(y=Total), stat="identity", fill="red") +
geom_bar(aes(y=Survived), stat="identity", fill="lightgreen") +
geom_text(aes(label = (round(Survived/Total,4) * 100)), color='blue', vjust = -5.25) +
xlab(col)
return(plot)
}
for (col in cols) {
vector[[col]] <- get_plot(full,col)
}
grid.arrange( grobs = vector, ncol = 2)
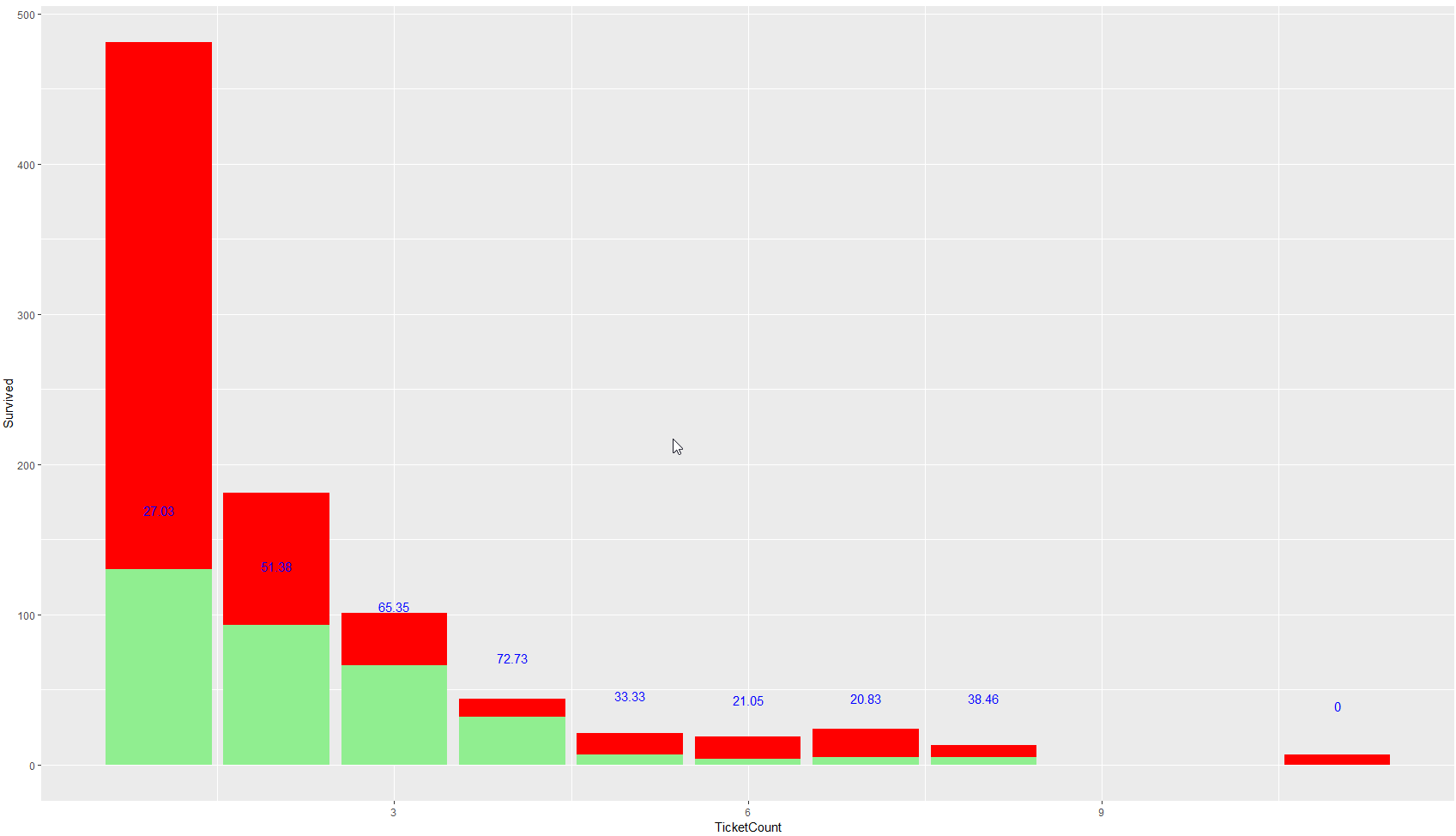
- Dodatkowo możemy obliczyć jakie jest prawdopodobieństwo przeżycia w zależności od osób które mają ten sam numer biletu.
get_plot(full,"TicketCount")
R / Uzupełnianie NaN
Embarked
Wpierw sprawdzamy jaka wartość jest pusta
full %>%
filter( is.na(Embarked) )

Z tego można wysnuć wniosek że można Embarked wypełnić wartościami z wierwszy o zbliżonych wartościach. Najpierw sprawdzimy korelacji wartości _Embarked od czego ona najbardziej zależy. Według danych zależy od _CabinType i Pclass
cols <- c('Age','Sex2', 'Embarked2','Fare', 'Parch' , 'Pclass', 'SibSp', 'Cabin2','TitleN')
abs(cor(full[,cols],use="complete.obs")[,"Embarked2"]) %>% .[order(., decreasing = TRUE)]
Embarked2 1
Cabin2 0.262454554103353
Fare 0.251148302194369
Pclass 0.227288284895879
TitleN 0.0905833350473123
Age 0.0856017843323628
SibSp 0.065656436575671
Parch 0.0596918664937904
Sex2 0.022812259698426
Tak więc potrzebujemy dla Pclass, Cabin2 obliczyć Fare i zobaczyć który Embarked jest najbliżej
full %>%
filter(Pclass == 1 & Cabin2 == 2 ) %>%
group_by(Embarked) %>%
summarise( sum = sum(Fare), count = n(), mean = mean(Fare), median = median(Fare))
Mogę także wyświetlić boxplot pomiędzy tymi wartościami aby zobaczyć która opłata jest bliższa naszej wartości. Na czerwonej linii zaznaczyłem wartość 80.0 (czyli opłata która jest tu wypełniona)
ggplot(data=full %>%filter(Pclass == 1 & Cabin2 == 2 & Embarked %in% c('C','S') ), aes(Embarked, Fare, group = Embarked)) +
geom_boxplot() + geom_hline(yintercept=80,color='red')
Wartości są bardzo zbliżone, i wartość 80 jest bliska zarówno miediany dla ‘C’jak i dla ‘S‘. Ponieważ jednak port ‘C’ma ceny bardziej rozsztrzelone mogę podejrzewać że portem źródłowym jest ‘S’(Southampton).
full[62,'Embarked'] = 'S'
full[830,'Embarked'] = 'S'
full$Embarked2 <- as.numeric(as.factor(full$Embarked))
Potrzeba także przeliczenia kolumny Embarked2 żeby zawierała także kody.
Fare
Dla Fare postąpiłem podobnie, znalazłem najbardziej skorelowane zmienne i wyznaczyłem średnie wartości.
full %>%
filter( is.na(Fare) )
cols <- c('Age','Sex2', 'Embarked2','Fare', 'Parch' , 'Pclass', 'SibSp', 'Cabin2','TitleN')
abs(cor(full[,cols],use="complete.obs")[,"Fare"]) %>% .[order(., decreasing = TRUE)]
Fare Parch Pclass Cabin2 SibSp Embarked2 Sex2 TitleN Age
1.00000000 0.39000359 0.31001901 0.30240349 0.26803086 0.25114830 0.14894854 0.08180847 0.01432193
full %>%
filter(Pclass == 3 & Embarked == 'S' & Parch == 0 & Sex =='male' & Age > 40) %>%
group_by(Age) %>%
summarise( sum = sum(Fare), count = n(), mean = mean(Fare), median = median(Fare))
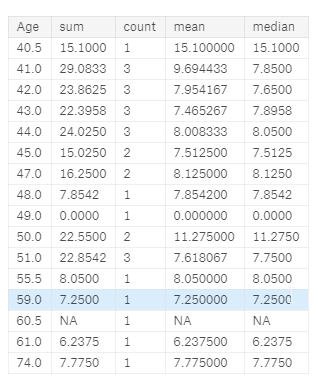
Można przypuszczać że osoby w tym wieku średnio płaciły między 6 a 7 funtów. Możemy w takim razie przyjąć średnią z tych dwóch wartości. Różnica między 6 a 7 jest zbyt mała aby mieć istotny wpływ w porównaniu do całego obszaru cen (od 0 do 512, gdzie średnia to 33 a mediana to 14)
full[1044,'Fare'] = (7.25 + 6.2375)/2 # we set average for this values
Age
Tutaj jest już spora ilość brakujących elementów, więc nie możemy jak poprzednio uzupełniać pojedyńcze wartości. Z pomocą przychodzi na.aggregate który uzupełnia domyślnie wartością średnią.
library(zoo)
full$Age <- as.vector((na.aggregate(full[,"Age"],na.rm=FALSE))$Age)
Cabin
Dla Cabin możemy użyć uzupełnienia przez najczęściej występujący element, ale nie chcemy używać elementu Cabin ponieważ jest za bardzo rozproszony. Dlatego użyjemy Cabin2.
val <- (full %>% drop_na(Cabin2) %>% count(Cabin2) %>% slice(which.max(n)) %>% select(Cabin2))$Cabin2
val
3
Puste wartości zostały wypełnione wartością 3.
full <- full %>% mutate(Cabin2 = ifelse( is.na(Cabin2),3, Cabin2) )
Można teraz wyświetlić sobie sumarycznie puste elementy. Nie licząc Survived, Cabin (które nie jest potrzebne), nie posiadamy już pustych wartości.
full %>%
select(everything()) %>% # replace to your needs
summarise_all(funs(sum(is.na(.))))
R/ Machine Learning
Teraz przyszedł czas na uczenie maszynowe i przepowiadanie wartości.
W tym wypadku trzeba podjąć 3 kroki:
- Znormalizować wszystkie kolumny biorące udział w liczeniu, w tym wypadku tylko kolumna Fare ponieważ kolumna Age została skategoryzowana.
- Wybrać najlepszy model na podstawie score wybrać model z najlepszym wynikiem.
- Przewidzieć wyniki i wysłać do sprawdzenia.
Normalizacja
Prawie wszystkie kolumny oprócz Fare są kolumnami kategorii. Fare warto zawsze przeskalować przed kalkulacjami tak żeby nie wpływał za bardzo na model.
full$Fare2 <- as.vector(scale(full$Fare))
Age Category
Warto także dodać AgeCategory który dzieli nam wiek na poszczególne kategorie. Ja podzieliłem go na 6 kategorii.
full$AgeCategory <- as.numeric(as.factor(cut(full$Age,breaks = c(0,9,18,30,40,50,100))))
Kolumny
Kolumny biorące udział w obliczeniach. Wszystkie kolumny są albo typu liczbowego albo kategorii. Powoduje to że mogą brać udział w klasyfikatorze.
cols <- c('Pclass','SibSp','Parch','Sex2','Embarked2','Cabin2','TitleN','TicketCount','AgeCategory','Fare2')
cols2 <- c('Pclass','SibSp','Parch','Sex2','Embarked2','Cabin2','TitleN','TicketCount','AgeCategory','Fare2','Survived')
full[,cols]
MLR - make task
Opis razem z rodzajami algorytmów można znaleść na stronie:
Machine Learning za pomocą paczki MLR wymaga wykonania tylko kilku operacji. Ma w sobie większość dostępnych algorytmów. Na początek spróbujemy xgboost czyli najpopularniejszy algorytm.
- MLR w Kaggle wymaga aby kolumna Survived była jako faktor, ponieważ jest to 0/1 zamieniamy ją na ten typ.
#ML Section
library(mlr)
#full_input <- normalizeFeatures(full_input, target = "Survived")
train_input <- full[,cols2] %>% filter(!is.na(Survived) )
train_input$Survived <- as.factor(train_input$Survived)
MakeClassificTask
Tutaj tworzymy zadanie dla MLR-a. Na zadaniu potem możemy go uczyć wieloma metodami, sprawdzać jak bardzo metody są dokładne. Nie ma tutaj podanego bezpośrednio algorytmu bo może być ich wiele. Najważniejszy jest typ zadania.
task = makeClassifTask(data = train_input, target = "Survived")
MakeLearner
Tutaj tworzymy ucznia (learner) do naszego zadania. Wybrałem najpowszechniejszy ‘classif.xgboost’.
xgb_learner <- makeLearner("classif.xgboost")
train
Trenujemy ucznia na podstawie danych z task.
mod = train(xgb_learner, task)
predict
Obliczamy jak dokładna jest nasza sieć w stosunku do trenowanych danych. (acc - jest najbardziej popularną miarą dokładności naszego modelu).
Wylicza się je prze dodanie wartości TP (TruePositve - Zgadnięte prawidłowo Survived = 1) i TN (True Negative - Zgadnięte jako Survived = 0 ale naprawdę powinno być 1) podzielone przez sumę wartośći Survived = 0 i Survived = 1.
pred = predict(mod, task = task)
print(performance(pred, measures = list("acc" = acc)))
acc
0.8698092
zapis do .csv
Aby zapisać do pliku .csv trzeba najpierw ściągnąć dane które chcemy przetestować, dodałem także kolumne PassengesID.
#predict
test_data <- full %>% filter(is.na(Survived) )
test_passengersID <- test_data[,c('PassengerId')]
test_input <- test_data[,cols]
Używamy jeszcze raz funkcji predict ale zamiast przewidywać na zadaniu, przewidujemy na newdata. Zwraca nam tylko rezultat.
pred <- as.data.frame(predict(mod,newdata = test_input))
Pozostaje wyniki zapisać do pliku .csv. cbind łączy kolumne PassengerID i Survived, colnames zmienia nazwę kolumny.
# write to csv
colnames(pred) = c("Survived")
write.csv(cbind(test_passengersID,pred),'output.csv', quote = FALSE, row.names = FALSE)
Wysłanie do Kaggle
Teraz wystarczy kliknąć na Notebook commit & run, a następnie przejść do output i znaleść (tutaj wykresy także pojawiają się w output) na końcu nasz plik output.csv i po kliknięciu “Submit to Compettion”powinien wyświetlić się wynik (u mnie 0.78). Nie jest do dużo, ale dane nie były szczególnie poprawianie.
R/ Końcowy kod
library(tidyverse) # metapackage with lots of helpful functions
list.files(path = "../input")
# csv read
train <- read_csv('../input/train.csv')
test <- read_csv('../input/test.csv')
# set full
test$Survived <- NA
full <- rbind(train, test)
# info
str(full)
dim(full)
nrow(full) #liczba kolumn
ncol(full) #liczba wierszy
head(full)
summary(full)
# Filter
library(dplyr)
full[,c("Survived","Pclass")]
select(full,Survived,Pclass)
full[1:9,]
filter(full, Age > 5.0 & Age < 7.0 )
full %>%
filter(Age > 5.0 & Age < 7.0 ) %>%
select(Survived,Pclass)
full %>%
filter( is.na(Fare) )
library(stringr)
full %>%
filter( str_detect(Cabin, 'B2') )
full %>%
drop_na(Survived) %>%
group_by(Pclass,Sex) %>%
summarise(Survived = sum(Survived))
full %>%
group_by(Pclass,Sex) %>%
summarise(Survived = sum(Survived, na.rm = T))
# Dane
full$Sex2 <- as.numeric(as.factor(full$Sex))
full$Embarked2 <- as.numeric(as.factor(full$Embarked))
full$Cabin2 <-as.numeric(as.factor(substring(full$Cabin, 0, 1) ))
cols <- c('Age','Sex2','Embarked2','Fare','Parch','Pclass','SibSp','Survived','Cabin2')
cor( full[,cols], use="complete.obs")
## Title
library(stringr)
full$Title <- str_match(full$Name, ",\\s([^ .]+)\\.?\\s+")[,2]
full %>%
group_by(Title) %>%
summarise(cnt = n()) %>%
arrange(desc(cnt))
full$Title2 <- full$Title
full$Title2[ full$Title %in% c('Mlle','Ms','Lady')] <- 'Miss'
full$Title2[ full$Title %in% c('Mme')] <- 'Mrs'
full$Title2[ full$Title %in% c('Sir')] <- 'Mr'
full$Title2[ ! full$Title %in% c('Miss','Master','Mr','Mrs')] <- 'Other'
full$TitleN <- as.numeric(as.factor(full$Title2))
full %>%
group_by(Title2) %>%
summarise(cnt = n()) %>%
arrange(desc(cnt))
## TicketCount
full <- full %>% group_by(Ticket) %>% mutate(TicketCount = n()) %>% ungroup()
# Wykresy
hist(full$Age)
hist(full$Age, breaks=60)
ggplot(data=full, aes(full$Age)) + geom_histogram()
ggplot(data=full, aes(full$Age)) + geom_histogram(bins=60)
ggplot(data=full, aes(x=Age)) +
geom_histogram(binwidth = 2.5, alpha = 0.2) +
geom_histogram(data = subset(full,Survived == 1), fill = "blue", alpha = 0.2) +
geom_density(aes(y=2.5 * ..count..),color="red",na.rm = T) +
geom_density(data = subset(full,Survived == 1), aes(y=2.5 * ..count..),color="blue",na.rm = T)
ggplot(data=full, aes(Survived, Age, group = Survived)) + geom_boxplot()
ggplot(data=full, aes(Pclass, Age, group = Pclass)) + geom_boxplot()
cols <- c('Age','Sex2','Embarked2','Fare','Parch','Pclass','SibSp','Survived','Cabin2')
corr <- cor( full[,cols], use="complete.obs")
heatmap(corr,
na.rm = T, # same data set for cell labels
)
library(reshape2)
corr_df <- melt(corr, na.rm = TRUE)
ggplot(corr_df, aes(x=Var1,y=Var2, fill=value)) +
geom_tile() +
geom_text(aes(Var2, Var1, label = round(value,2)), color = "white", size = 4)
# install.packages("PerformanceAnalytics")
library("PerformanceAnalytics")
cols <- c('Age','Sex2','Embarked2','Fare','Parch','Pclass','SibSp','Survived','Cabin2','TitleN')
chart.Correlation(full[,cols], histogram=TRUE, pch=19, font.size = 15)
# Grupowe analizy
library(ggplot2)
require(gridExtra)
full$AgeCut <- cut(full$Age,breaks = seq(0, 100, by = 10))
full$FareCut <- cut(full$Fare,10)
cols <- c('AgeCut','Sex','Embarked','FareCut','Parch','Pclass','SibSp', 'Cabin2','Title2')
vector <- list()
get_plot <- function (data, col) {
data_group <- data[,c(col,"Survived")] %>%
select(x = col, "Survived") %>%
drop_na(Survived) %>%
group_by(x) %>%
summarise(Total = n(),Survived = sum(Survived,na.rm = T), ratio = sum(Survived,na.rm = T)/n())
plot <- ggplot(data_group ,aes(x, Survived, label='A')) +
geom_bar(aes(y=Total), stat="identity", fill="red") +
geom_bar(aes(y=Survived), stat="identity", fill="lightgreen") +
geom_text(aes(label = (round(Survived/Total,4) * 100)), color='blue', vjust = -5.25) +
xlab(col)
return(plot)
}
for (col in cols) {
vector[[col]] <- get_plot(full,col)
}
grid.arrange( grobs = vector, ncol = 2)
get_plot(full,"TicketCount")
# Uzupełnianie NaN
## Embarked
full %>%
filter( is.na(Embarked) )
cols <- c('Age','Sex2', 'Embarked2','Fare', 'Parch' , 'Pclass', 'SibSp', 'Cabin2','TitleN')
abs(cor(full[,cols],use="complete.obs")[,"Embarked2"]) %>% .[order(., decreasing = TRUE)]
full %>%
filter(Pclass == 1 & Cabin2 == 2 ) %>%
group_by(Embarked) %>%
summarise( sum = sum(Fare), count = n(), mean = mean(Fare), median = median(Fare))
ggplot(data=full %>%filter(Pclass == 1 & Cabin2 == 2 & Embarked %in% c('C','S') ), aes(Embarked, Fare, group = Embarked)) +
geom_boxplot() + geom_hline(yintercept=80,color='red')
full[62,'Embarked'] = 'S'
full[830,'Embarked'] = 'S'
full$Embarked2 <- as.numeric(as.factor(full$Embarked))
## Fare
full %>%
filter( is.na(Fare) )
cols <- c('Age','Sex2', 'Embarked2','Fare', 'Parch' , 'Pclass', 'SibSp', 'Cabin2','TitleN')
abs(cor(full[,cols],use="complete.obs")[,"Fare"]) %>% .[order(., decreasing = TRUE)]
full %>%
filter(Pclass == 3 & Embarked == 'S' & Parch == 0 & Sex =='male' & Age > 40) %>%
group_by(Age) %>%
summarise( sum = sum(Fare), count = n(), mean = mean(Fare), median = median(Fare))
full[1044,'Fare'] = (7.25 + 6.2375)/2 # we set average for this values
## Age
library(zoo)
full$Age <- as.vector((na.aggregate(full[,"Age"],na.rm=FALSE))$Age)
## Cabin
val <- (full %>% drop_na(Cabin2) %>% count(Cabin2) %>% slice(which.max(n)) %>% select(Cabin2))$Cabin2
val
full <- full %>% mutate(Cabin2 = ifelse( is.na(Cabin2),3, Cabin2) )
## Summarise N/A
full %>%
select(everything()) %>% # replace to your needs
summarise_all(funs(sum(is.na(.))))
# Normalizacja
full$Fare2 <- as.vector(scale(full$Fare))
# Age categorize
full$AgeCategory <- as.numeric(as.factor(cut(full$Age,breaks = c(0,9,18,30,40,50,100))))
cols <- c('Pclass','SibSp','Parch','Sex2','Embarked2','Cabin2','TitleN','TicketCount','AgeCategory','Fare2')
cols2 <- c('Pclass','SibSp','Parch','Sex2','Embarked2','Cabin2','TitleN','TicketCount','AgeCategory','Fare2','Survived')
full[,cols]
full[,cols2]
#ML Section
library(mlr)
#full_input <- normalizeFeatures(full_input, target = "Survived")
train_input <- full[,cols2] %>% filter(!is.na(Survived) )
train_input$Survived <- as.factor(train_input$Survived)
# Create an xgboost learner that is classification based and outputs
#install.packages("kernlab")
task = makeClassifTask(data = train_input, target = "Survived")
xgb_learner <- makeLearner("classif.xgboost")
mod = train(xgb_learner, task)
pred = predict(mod, task = task)
print(performance(pred, measures = list("acc" = acc)))
#predict
test_data <- full %>% filter(is.na(Survived) )
test_passengersID <- test_data[,c('PassengerId')]
test_input <- test_data[,cols]
pred <- as.data.frame(predict(mod,newdata = test_input))
# write to csv
colnames(pred) = c("Survived")
write.csv(cbind(test_passengersID,pred),'output.csv', quote = FALSE, row.names = FALSE)
R/ Ulepszanie modelu
Wynik który uzyskaliśmy dla takiego zadania da się na pewno poprawić. na Kaggle nawet można zobaczyć wyniki wynoszące 1.0. Taki wynik może znaczyć że użytkownicy prawdopodobnie nie używali modelu a mają już zapisane wyniki które wcześniej przetestowali ponieważ prawie niemożliwością jest za pomocą modeli uzyskanie idealnego wyniku.
Na początku może zobaczymy na kod z którego usunąłem niepotrzebne elementy.
library(tidyverse) # metapackage with lots of helpful functions
list.files(path = "../input")
# csv read
train <- read_csv('../input/train.csv')
test <- read_csv('../input/test.csv')
# set full
test$Survived <- NA
full <- rbind(train, test)
# Dane
full$Sex2 <- as.numeric(as.factor(full$Sex))
full$Embarked2 <- as.numeric(as.factor(full$Embarked))
full$Cabin2 <-as.numeric(as.factor(substring(full$Cabin, 0, 1) ))
## Title
full$Title <- str_match(full$Name, ",\\s([^ .]+)\\.?\\s+")[,2]
full$Title2 <- full$Title
full$Title2[ full$Title %in% c('Mlle','Ms','Lady')] <- 'Miss'
full$Title2[ full$Title %in% c('Mme')] <- 'Mrs'
full$Title2[ full$Title %in% c('Sir')] <- 'Mr'
full$Title2[ ! full$Title %in% c('Miss','Master','Mr','Mrs')] <- 'Other'
full$TitleN <- as.numeric(as.factor(full$Title2))
## TicketCount
full <- full %>% group_by(Ticket) %>% mutate(TicketCount = n()) %>% ungroup()
# Uzupelnianie NaN
## Embarked
full[62,'Embarked'] = 'S'
full[830,'Embarked'] = 'S'
full$Embarked2 <- as.numeric(as.factor(full$Embarked))
## Fare
full[1044,'Fare'] = (7.25 + 6.2375)/2 # we set average for this values
## Age
library(zoo)
full$Age <- as.vector((na.aggregate(full[,"Age"],na.rm=FALSE))$Age)
## Cabin
full <- full %>% mutate(Cabin2 = ifelse( is.na(Cabin2),3, Cabin2) )
## Summarise N/A
# full %>% select(everything()) %>% summarise_all(funs(sum(is.na(.))))
# Normalizacja
full$Fare2 <- as.vector(scale(full$Fare))
# Age categorize
full$AgeCategory <- as.numeric(as.factor(cut(full$Age,breaks = c(0,9,18,30,40,50,100))))
cols <- c('Pclass','SibSp','Parch','Sex2','Embarked2','Cabin2','TitleN','TicketCount','AgeCategory','Fare2')
cols2 <- c('Pclass','SibSp','Parch','Sex2','Embarked2','Cabin2','TitleN','TicketCount','AgeCategory','Fare2','Survived')
#ML Section
library(mlr)
#full_input <- normalizeFeatures(full_input, target = "Survived")
train_input <- full[,cols2] %>% filter(!is.na(Survived) )
train_input$Survived <- as.factor(train_input$Survived)
# Create an xgboost learner that is classification based and outputs
#install.packages("kernlab")
task = makeClassifTask(data = train_input, target = "Survived")
xgb_learner <- makeLearner("classif.xgboost")
mod = train(xgb_learner, task)
pred = predict(mod, task = task)
print(performance(pred, measures = list("acc" = acc)))
#predict
test_data <- full %>% filter(is.na(Survived) )
test_passengersID <- test_data[,c('PassengerId')]
test_input <- test_data[,cols]
pred <- as.data.frame(predict(mod,newdata = test_input))
# write to csv
colnames(pred) = c("Survived")
write.csv(cbind(test_passengersID,pred),'output.csv', quote = FALSE, row.names = FALSE)
Automatyzacja wysyłania na kaggle:
LIMITY
Kaggle ma limity na wysyłanie odpowiedzi do 10 w ciągu dnia. Dlatego radze mieć to na uwadze.
Wysyłanie wyników na serwer przez stronę Kaggle jest uciążliwe, dlatego istnieje automat do wysyłania naszych wyników.
Wystarczy zainstalować kaggle:
pip install kaggle
Wrzucić nasz API-Key do konfiguracji użytkownika:
https://github.com/Kaggle/kaggle-api
I można wygodnie wysyłać z konsoli nasze wyniki
kaggle competitions submit -c titanic -f submission.csv -m "Message"
Na stronię https://www.kaggle.com/c/titanic/submissions mamy możliwość przejrzenia naszych wyników.
Na początku mamy wynik 0.78468:

Od tej chwile będe używał lokalnego kodu i sprawadzał wyniki bezpośrednio.
Co można poprawić w takim kodzie?
Imputer wpisuje jedną wartość dla wszystkich identyczną, fajnie by było gdyby można było wyznaczyć wiek na podstawie innych kolumn.
Można sparametryzować XGBoost i wyszukać najlepsze parametry dla lepszego dopasowania
Poprawianie pustych wartości dla Age
Wartości Age ustawiłem na jeden z góry zadaną wartość. Może można poprawić przez korelacje z innymi zmiennymi.
abs(cor(full[,c('Age','Sex2', 'Embarked2','Fare', 'Parch' , 'Pclass', 'SibSp', 'Cabin2','TitleN')],use="complete.obs")[,"Age"]) %>% .[order(., decreasing = TRUE)]
Age TitleN Pclass SibSp Fare Parch Embarked2 Sex2 Cabin2
1.00000000 0.47146342 0.36637081 0.19074677 0.17057145 0.13087166 0.07118146 0.05739710 0.05589489
Korelacja silnie zależy od tytułu i Pclass. Tak więc można użyć mediany dla odpowiadających im tytułów. Znajduje się jedna pusta wartości dal TitleN = 5 i Pclass = 3, tak więc uzupełniamy ją wartością dla TitleN = 5.
## Age
# abs(cor(full[,c('Age','Sex2', 'Embarked2','Fare', 'Parch' , 'Pclass', 'SibSp', 'Cabin2','TitleN')],use="complete.obs")[,"Age"]) %>% .[order(., decreasing = TRUE)]
title.age <- aggregate(full$Age,by = list(full$TitleN,full$Pclass), FUN = function(x) median(x, na.rm = T))
title.age[15,]$x <- 41
full[is.na(full$Age), "Age"] <- apply(full[is.na(full$Age), ] , 1, function(x) title.age[title.age[, 1]==x["TitleN"] & title.age[, 2]==x["Pclass"], 3])
# Age categorize
full$AgeCategory <- as.numeric(as.factor(cut(full$Age,breaks = c(0,9,18,30,40,50,100))))
Po wysłaniu:
Model poprawił się nieznacznie do 0.78947
Poprawianie MLR
Pierwsze co zrobiłem to zamiast zwykłej normalizacji użyłem normalizacji z MLR przez normalizeFeatures.
#ML Section
library(mlr)
cols <- c('Pclass','SibSp','Parch','Sex2','Embarked2','Cabin2','TitleN','TicketCount','AgeCategory','Fare')
cols2 <- c('Pclass','SibSp','Parch','Sex2','Embarked2','Cabin2','TitleN','TicketCount','AgeCategory','Fare','Survived')
full[,cols2] <- normalizeFeatures(full[,cols2], target = "Survived")
Do tego możemy używać hyperparametrów, czyli dodać parametry do naszego algorytmu xgboost par.vals a następnie za pomocą makeParamSet ustawić dolne i górne wartości parametru, algorytm sam wyszuka najlepsze parametry i które można użyć jako $x. makeTuneControlRandom służy do kontroli iteracji po ilu ma się zakończyć. makeResampleDesc określa w jaki sposób dane zostaną podzielone i obliczony score dla wyniku. Użyte została Cross Validation co pozwoliło podzielić test na 4 grupy. W każdej grupie model jest trenowane na 3 częściach a validacja na 4 części. Parametrami posłużyłem się z innego Kaggle.
https://www.kaggle.com/dkyleward/xgboost-using-mlr-package-r
Szczegóły na temat Cross Validation można znaleść w artykule:
https://medium.com/@mtterribile/understanding-cross-validations-purpose-53490faf6a86
| Training set | |
| Test set (Validation set) |
# getParamSet("classif.xgboost")
xgb_learner <- makeLearner(
"classif.xgboost",
predict.type = "response",
par.vals = list(
objective = "binary:logistic",
eval_metric = "error",
nrounds = 200
)
)
par.set <- makeParamSet(
# The number of trees in the model (each one built sequentially)
makeIntegerParam("nrounds", lower = 100, upper = 500),
# number of splits in each tree
makeIntegerParam("max_depth", lower = 1, upper = 10),
# "shrinkage" - prevents overfitting
makeNumericParam("eta", lower = .1, upper = .5),
# L2 regularization - prevents overfitting
makeNumericParam("lambda", lower = -1, upper = 0, trafo = function(x) 10^x)
)
control = makeTuneControlRandom(maxit = 20)
# Create a description of the resampling plan
resampling <- makeResampleDesc("CV", iters = 4) #Cross Validation
tuned_params <- tuneParams(
learner = xgb_learner,
task = task,
resampling = resampling,
par.set = par.set,
control = control
)
# Create a new model using tuned hyperparameters
xgb_tuned_learner <- setHyperPars(
learner = xgb_learner,
par.vals = tuned_params$x
)
mod = train(xgb_tuned_learner, task)
pred = predict(mod, task = task)
print(performance(pred, measures = list("acc" = acc)))
Co uzyskaliśmy:
0.95 w xgboost, natomiast w Kaggle dla testu: 0.73
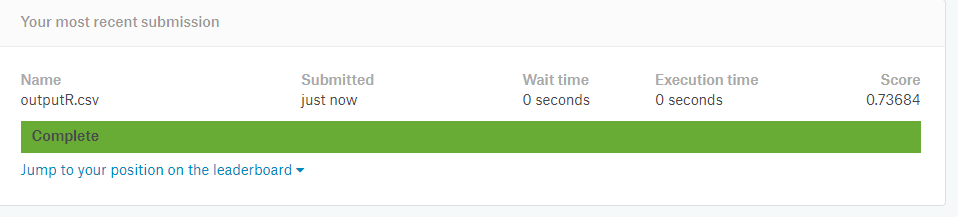
Jak się okazało polepszenie sieci wcale nie polepszyło wyniku a nawet go pogorszyło.
Co spowodowało taki spadek, a mianowicie przeuczenie modelu. XGBoost uzyskał tak dobry wynik dla danych treningowych że model nie poradził sobie z danymi testowymi tak dobrze, jak gdy nie dopasowała się aż tak dobrze poprzednio (miało odpowiednio score: 0.884 dla treningowych i 0.78 dla testu). Co obrazuje poniższy obrazek i przykład wzięty z uczenia sieci neuronowej.

źródło: https://www.quora.com/Is-it-necessary-to-get-rid-of-useless-features-in-the-classification-task-in-XGBoost
źródło: http://www.grandjean-bpa.com/UL-nnfit/english/man/nnfit2_man.html
Jak widać ten model idealnie dopasował wartości ale czy dla wartości które nie zna jest aż tak dobre. Okazuje się że nie.
MLR/2
Jednym z metod jest użycie Cross-Validation, ale już użyłem go w poprzednim i nie poprawiło to dobrze algorytmu. Do poprawy użyłem kolejnych parametrów zgodnie z radami na stronie XGBoost.
http://xgboost.readthedocs.io/en/latest/how_to/param_tuning.html
par.set <- makeParamSet(
# The number of trees in the model (each one built sequentially)
makeIntegerParam("nrounds", lower = 100, upper = 500),
# number of splits in each tree
makeIntegerParam("max_depth", lower = 1, upper = 10),
# "shrinkage" - prevents overfitting
makeNumericParam("eta", lower = .1, upper = .5),
# L2 regularization - prevents overfitting
makeNumericParam("lambda", lower = -1, upper = 0, trafo = function(x) 10^x),
# Dla Overfitting
makeNumericParam("gamma", -15, 15, trafo = function(x) 2^x),
makeNumericParam("subsample", lower = 0.10, upper = 0.80),
makeNumericParam("min_child_weight",lower=1,upper=5),
makeNumericParam("colsample_bytree",lower = 0.2,upper = 0.8)
)
Oraz dodałem resample() który dopasowuje model dla zadania przez podział modelu na kawałki i testowanie każdego kawałka z osobna. w ResampleDesc opisałem model Cross-Validation przedstawiony wcześniej. Wartości sprawdzane dla modelu to acc i mmce.
# Verify performance on cross validation folds of tuned model
resampling <- makeResampleDesc("CV", iters = 4) #Cross Validation
resample(xgb_tuned_learner,task,resampling,measures = list(acc,mmce))
Wyniki przestawiają się następująco.
Resampling: cross-validation
Measures: acc mmce
[Resample] iter 1: 0.8475336 0.1524664
[Resample] iter 2: 0.8251121 0.1748879
[Resample] iter 3: 0.8116592 0.1883408
[Resample] iter 4: 0.7972973 0.2027027
Aggregated Result: acc.test.mean=0.8204006,mmce.test.mean=0.1795994
Cały kod przedstawia się następująco:
library(tidyverse) # metapackage with lots of helpful functions
list.files(path = "../input")
# csv read
train <- read_csv('../input/train.csv')
test <- read_csv('../input/test.csv')
# set full
test$Survived <- NA
full <- rbind(train, test)
# Dane
full$Sex2 <- as.numeric(as.factor(full$Sex))
full$Embarked2 <- as.numeric(as.factor(full$Embarked))
full$Cabin2 <-as.numeric(as.factor(substring(full$Cabin, 0, 1) ))
## Title
full$Title <- str_match(full$Name, ",\\s([^ .]+)\\.?\\s+")[,2]
full$Title2 <- full$Title
full$Title2[ full$Title %in% c('Mlle','Ms','Lady')] <- 'Miss'
full$Title2[ full$Title %in% c('Mme')] <- 'Mrs'
full$Title2[ full$Title %in% c('Sir')] <- 'Mr'
full$Title2[ ! full$Title %in% c('Miss','Master','Mr','Mrs')] <- 'Other'
full$TitleN <- as.numeric(as.factor(full$Title2))
## TicketCount
full <- full %>% group_by(Ticket) %>% mutate(TicketCount = n()) %>% ungroup()
# Uzupelnianie NaN
## Embarked
full[62,'Embarked'] = 'S'
full[830,'Embarked'] = 'S'
full$Embarked2 <- as.numeric(as.factor(full$Embarked))
## Fare
full[1044,'Fare'] = (7.25 + 6.2375)/2 # we set average for this values
## Age
# abs(cor(full[,c('Age','Sex2', 'Embarked2','Fare', 'Parch' , 'Pclass', 'SibSp', 'Cabin2','TitleN')],use="complete.obs")[,"Age"]) %>% .[order(., decreasing = TRUE)]
title.age <- aggregate(full$Age,by = list(full$TitleN,full$Pclass), FUN = function(x) median(x, na.rm = T))
title.age[15,]$x <- 41
full[is.na(full$Age), "Age"] <- apply(full[is.na(full$Age), ] , 1, function(x) title.age[title.age[, 1]==x["TitleN"] & title.age[, 2]==x["Pclass"], 3])
# Age categorize
full$AgeCategory <- as.numeric(as.factor(cut(full$Age,breaks = c(0,9,18,30,40,50,100))))
## Cabin
full[is.na(full$Cabin2), "Cabin2"] <- 3
## Summarise N/A
# full %>% select(everything()) %>% summarise_all(funs(sum(is.na(.))))
#ML Section
library(mlr)
cols <- c('Pclass','SibSp','Parch','Sex2','Embarked2','Cabin2','TitleN','TicketCount','AgeCategory','Fare')
cols2 <- c('Pclass','SibSp','Parch','Sex2','Embarked2','Cabin2','TitleN','TicketCount','AgeCategory','Fare','Survived')
# Normalizacja
# full$Fare2 <- as.vector(scale(full$Fare))
full[,cols2] <- normalizeFeatures(full[,cols2], target = "Survived")
#full_input <- normalizeFeatures(full_input, target = "Survived")
train_input <- full[,cols2] %>% filter(!is.na(Survived) )
train_input$Survived <- as.factor(train_input$Survived)
# Create an xgboost learner that is classification based and outputs
#install.packages("kernlab")
task = makeClassifTask(data = train_input, target = "Survived")
# getParamSet("classif.xgboost")
xgb_learner <- makeLearner(
"classif.xgboost",
predict.type = "response",
par.vals = list(
objective = "binary:logistic",
eval_metric = "error",
nrounds = 200
)
)
par.set <- makeParamSet(
# The number of trees in the model (each one built sequentially)
makeIntegerParam("nrounds", lower = 100, upper = 500),
# number of splits in each tree
makeIntegerParam("max_depth", lower = 1, upper = 10),
# "shrinkage" - prevents overfitting
makeNumericParam("eta", lower = .1, upper = .5),
# L2 regularization - prevents overfitting
makeNumericParam("lambda", lower = -1, upper = 0, trafo = function(x) 10^x),
# Dla Overfitting
makeNumericParam("gamma", -15, 15, trafo = function(x) 2^x),
makeNumericParam("subsample", lower = 0.10, upper = 0.80),
makeNumericParam("min_child_weight",lower=1,upper=5),
makeNumericParam("colsample_bytree",lower = 0.2,upper = 0.8)
)
control = makeTuneControlRandom(maxit = 20)
# Create a description of the resampling plan
resampling <- makeResampleDesc("CV", iters = 4) #Cross Validation
tuned_params <- tuneParams(
learner = xgb_learner,
task = task,
resampling = resampling,
par.set = par.set,
control = control
)
# Create a new model using tuned hyperparameters
xgb_tuned_learner <- setHyperPars(
learner = xgb_learner,
par.vals = tuned_params$x
)
# Verify performance on cross validation folds of tuned model
resample(xgb_tuned_learner,task,resampling,measures = list(acc,mmce))
mod = train(xgb_tuned_learner, task)
pred = predict(mod, task = task)
print(performance(pred, measures = list("acc" = acc)))
#predict
test_data <- full %>% filter(is.na(Survived) )
test_passengersID <- test_data[,c('PassengerId')]
test_input <- test_data[,cols]
pred <- as.data.frame(predict(mod,newdata = test_input))
# write to csv
colnames(pred) = c("Survived")
write.csv(cbind(test_passengersID,pred),'outputR.csv', quote = FALSE, row.names = FALSE)
Po wysłaniu go do Kaggle dostajemy wynik 0.77033:
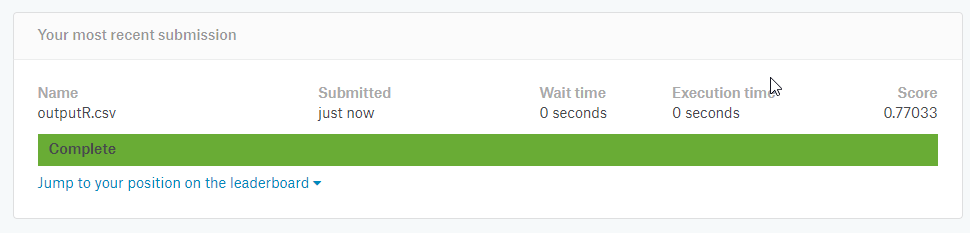
Warto poeksmerynteować z parametrami i sprawdzić czy nie da się jeszcze czegoś zrobić, albo jeszcze dodatkowo zmienić niektóre kolumny albo dodatkowo wyciągnąć dodatkowe dane. Warto poeksperymentować 😃
Przykład pojedyńczego Drzewa Decyzyjnego
Na końcu chciaem przedstawić przykład który został do mnie nadesłany przez Pawła Kaspierkiewicza w którym wykonał obliczenia za pomocą jednego drzewa decyzyjnego z biblioteki rpart (https://www.rdocumentation.org/packages/rpart/versions/4.1-13/topics/rpart ). Wyniki jakie uzyskał są bardzo dobre i wyglądają bardzo dobrze jak nie lepiej w porównaniu do XGBoost 😄
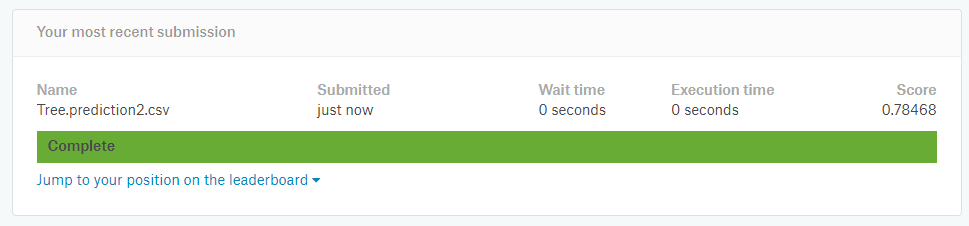
Pobieranie danych
#Fetch data
Train <- read.csv("Titanic.train.proc1.csv")
Train$X <- NULL
Train$Survived <- as.factor(Train$Survived)
Train$PassengerId <- NULL
Train$Name <- NULL
Train$Ticket <- as.factor(Train$Ticket)
Train$Pclass <- as.factor(Train$Pclass)
Data.to.predict <- read.csv("Titanic.test.proc1.csv")
Data.to.predict$Ticket <- as.factor(Data.to.predict$Ticket)
Data.to.predict$Pclass <- as.factor(Data.to.predict$Pclass)
Sprawdzanie korelacji
#Data analysis
#Yule.cor - my function for calculate Yule coefficient
library(MyLibrary) #My functions library
#Pclass
Yule.cor(Train$Survived, Train$Pclass)
table(Train$Survived, Train$Pclass)
| 1 | 2 | 3 | |
|---|---|---|---|
| 0 | 80 | 97 | 372 |
| 1 | 136 | 87 | 119 |
#Ticket
Yule.cor(Train$Survived, Train$Ticket)
table(Train$Survived, Train$Ticket)
| 0 | 1 | |
|---|---|---|
| 0 | 407 | 142 |
| 1 | 254 | 88 |
#Cabin
Yule.cor(Train$Survived, Train$Cabin)
table(Train$Survived, Train$Cabin)
| A | Other | |
|---|---|---|
| 0 | 68 | 481 |
| 1 | 136 | 206 |
#Embarked
Yule.cor(Train$Survived, Train$Embarked)
table(Train$Survived, Train$Embarked)
| C | Q | S | |
|---|---|---|---|
| 0 | 75 | 47 | 427 |
| 1 | 93 | 30 | 219 |
#Family
Yule.cor(Train$Survived, Train$Family)
table(Train$Survived, Train$Family)
| 0 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 10 | |
|---|---|---|---|---|---|---|---|---|---|
| 0 | 374 | 72 | 43 | 8 | 12 | 19 | 8 | 6 | 7 |
| 1 | 163 | 89 | 59 | 21 | 3 | 3 | 4 | 0 | 0 |
#Fare
Fare.surv <- Train[Train$Survived == 1, "Fare"]
Fare.dead <- Train[Train$Survived == 0, "Fare"]
summary(Fare.dead)
summary(Fare.surv)
| Min | 1st Qu. | Median | Mean | 3rd Qu. | Max. | |
|---|---|---|---|---|---|---|
| Fare.dead | 0.000 | 7.854 | 10.500 | 22.118 | 26.000 | 263.000 |
| Fare.surv | 0.000 | 12.47 | 26.00 | 48.40 | 57.00 | 512.33 |
hist(Fare.dead, xlim = c(0, 500), ylim = c(0, 300), breaks = 15)
hist(Fare.surv, xlim = c(0, 500), ylim = c(0, 300), breaks = 15)
boxplot(Fare.dead, Fare.surv)
plot(Fare.dead)
plot(Fare.surv)
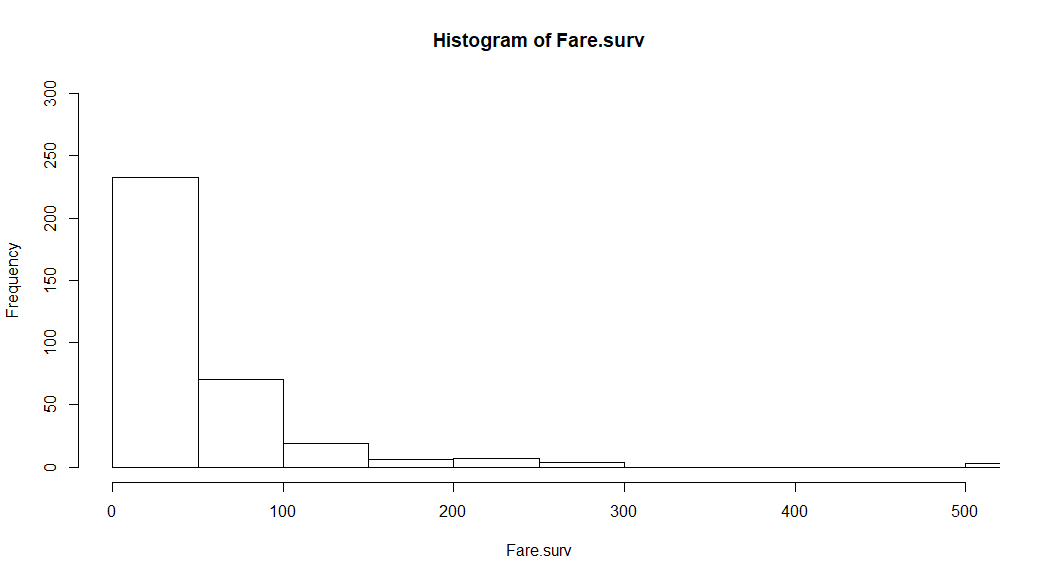
#Tempering outlier values
Train$Fare[Train$Fare > 300] <- 263
#Ticket and Embarked have less Yule's coefficient, so I removed it from model
Train$Ticket <- NULL
Train$Embarked <- NULL
Zbudowanie modelu
Funkcja rpart buduje nam drzewo decyzyjne.
library(rpart)
library(rattle)
set.seed(6)
Tree.1 <- rpart(Survived ~ ., cp = 0, minsplit = 2, data = Train, method = "class", xval = 300)
Dzięki plotcp możemy zobaczyć przy jakim poziomie przecięciu drzewa otrzymamy najmniejszy błąd.
https://www.rdocumentation.org/packages/rpart/versions/4.1-13/topics/plotcp
cp to Complexity Parameter in Decision Tree które pozwala wybrać optymalną wielkość drzewa decyzyjnego.
A za pomocą biblioteki rattle możemy zobaczyć jak wygląda drzewo decyzyjne.
Teraz wybieramy optymalną wartość CP tak żeby dostać jak najmniejszy błąd
CP <- Tree.1$cptable
CP <- as.data.frame(CP)
#Min cv error method for selecting CP
attach(CP)
optim.CP <- CP[CP$xerror == min(xerror), ]
optim.CP <- optim.CP[1, ]
detach(CP)
optim.CP <- as.numeric(optim.CP[1])
optim.CP # 0.002192982,
0.002192982
Przycinamy drzewo przez funkcje prune
#Submin CV error method for selecting CP
optim.CP <- CP[5, 1] # CP = 0.0073099415
#Pruning tree with submin CV CP value
Tree.1.2 <- prune(Tree.1, cp = optim.CP)
library(rattle)
fancyRpartPlot(Tree.1.2, cex = 0.6)
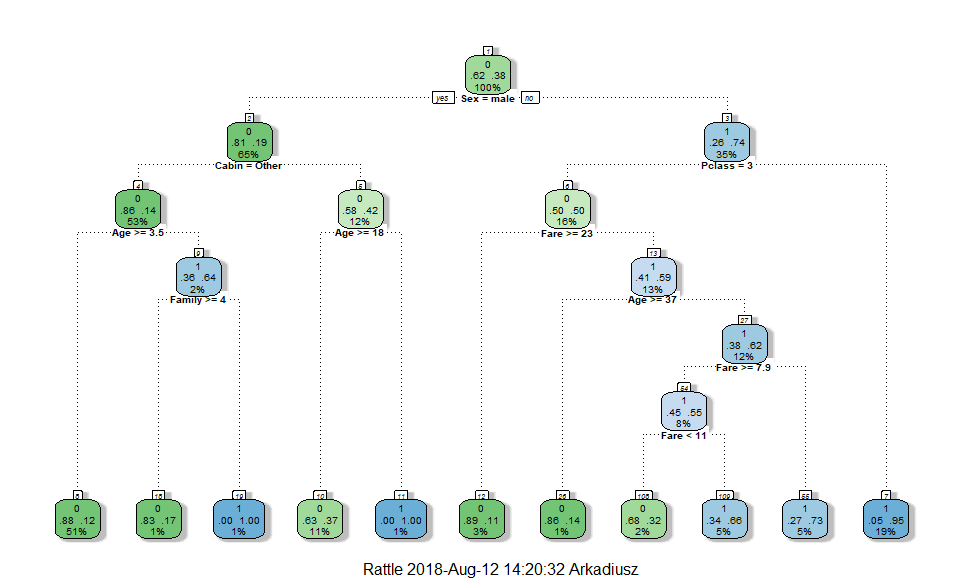
Przewidujemy za pomocą modelu wynik i zapisujemy go do .csv
predict.Tree.1.2 <- predict(Tree.1.2, type = "class")
sum(predict.Tree.1.2 == Train$Survived)/nrow(Train)
#Prediction and create answer table
Answer <- data.frame(PassengerId = Data.to.predict$PassengerId, Survived = predict(Tree.1.2, newdata = Data.to.predict, type = "class"))
rite.csv(Answer, "Tree.prediction2.csv", quote = FALSE, row.names = FALSE)
Wynik lokalny jaki uzyskujemy to 0.843, natomiast w Kaggle dostajemy 0.78468
0.8428732